自分で「とても分かりやすく」って言うかよ(笑)、って気もしましたが、書いてしまった以上、とても分かりやすくなるように全力を尽くすとしましょう。
「DNAの読み方」といっても「ディー・エヌ・エー。日本語でデオキシリボ核酸のこと」とかそういう話ではなく、DNAはA, C, G, Tとう4種類の構成単位(ヌクレオチドともいうし、塩基ともいう。100文字から成るDNAは、「100塩基のDNA」みたいな感じで)がズラーッとつながってできているという話でしたが、その並び方=文字の順番の読み方・調べ方の話ということですね。
前回、クッソしょうもない、ノーベル賞を複数回受賞したおっちゃんズの話とともに、「配列を読むことを、シークエンシング(シーケンシング)という」などと書いていましたが、一口にDNAシーケンシングといっても、様々なテクニックが開発・改良され、今に至っているのです、なんてことも既に書いていました。
今回は、そんな様々な手法の中から、今現在の生命科学研究の現場で恐らく最もよく使われている技法、サンガー法(を改良した手法)を紹介するとしましょう。
まず必要となる知識ですが……あんまり細かいことを書くと、途端にとても分かりやすくなくなってしまう気もするんですけど、最低限どうしても必要な話として、以下の点が挙げられます。
・DNAはヌクレオチド(より正確にはデオキシヌクレオチド)が連なった長い鎖だが、二本の鎖が互いに手をつなぐことで二本鎖を形成している。
・その「手のつなぎ方」は、全塩基が相棒の塩基と手をつないでいる形……つまり、A⇔T、C⇔Gが手をつないでいる。なので、一方の鎖の配列が分かれば、自動的に、相手の鎖の配列も確定する。
・しかも、DNAの鎖には向きがある。5'→3'として書き表される方向で、DNAの鎖が伸びる(新しいヌクレオチドが付加されて伸長していく)のはこの向きになる。
・しかも×2、二本鎖は、必ず、5'→3'という向きの鎖と、3'←5'という向き(180°逆向きの鎖)に並んで手をつないでいる形になっている。
…ってな所が、シーケンスについて語る上で外せない知識かと思います。
言葉だけでは分かりにくいですね。
今回も図を出して説明する予定ですが、いくつか図を貼って解説していた以前の記事も参考になるかもしれません。
(向きも大事!とか、DNAとRNAの違いをおさえておこうとか、割と最近のこの辺の記事・図を見れば一発?!改めて、分かりやすくDNA切り貼りの仕組みを紹介!とか、その辺で書いた話も……と思って記事を見直してみたら(特に「向きも大事!」とか)、書き上げた直後は「う~ん、分かりやすく書けたぞ」とか思ってた記憶があるのに、時間が経って改めて読んでみると、クッソ分かり辛いにも程があってたまげましたね。
あまりにもアレだったのでちょろっと修正しておきましたが、まぁ以前の記事はともかく、今回改めて分かりやすく説明してみようと思います。)
まずざっくりいうと、サンガー法の肝は、「ジデオキシヌクレオチドを用いる」という点に尽きるといえましょう。
いきなり分かりづらいカタカナで気分がムカムカするかもしれませんが、よく見たらこれはデオキシヌクレオチド、つまりDNAの構成分子であるdA, dC, dG, dTに、「ジ」がついただけなんですね。
つまり、de・oxy(無・酸素)で「酸素なし」という意味でしたが、ここにギリシャ語で「2」を意味するジがついただけなので、酸素がさらにもう1つなくなったデオキシヌクレオチドという、それだけに過ぎません。
先ほどリンクを貼ったDNAとRNAの違いをおさえておこうの記事でも使っていた図ですが、こちらがヌクレオチド(とデオキシヌクレオチド)の構造ですね。
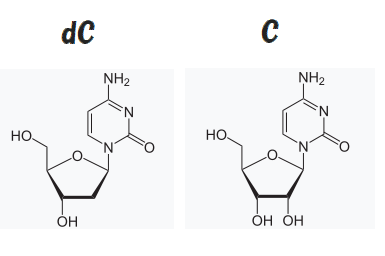
思えばこの図を貼ったことから、唐突に「分かりやすい有機化学講座」が始まったんでしたが、その講座で散々見てきたとおり、dC(=DNAのC)とC(=RNAのC)の構造を見比べますと、一箇所、OHがなくなっていることがお分かりになるかと思います(Hは図からは省略されているので、実際にはOがなくなっているだけ=デオキシということですね)。
そしてdCにはまだあと2つ、Oを頂点とする五角形の左下と左上にOHが残っているんですけど、この内の1つ、左下のOHをさらになくしたのが、「ジ・デオキシ・ヌクレオチド」っていうことなわけです。
ちなみに、Oを頂点として、そこから時計回りに、五角形の頂点の炭素(炭素原子は、図からは省略されていますが)を1'番、2'番、…、5'番(最後の5'番は、五角形の左上にはみ出た部分ですが)となっており、DNAの方向の5'→3'というのは何を隠そう、この番号のことなんですね。
つまり、この画像はヌクレオチド1分子ですが、5'のOHと3'のOHが手をつなぐことで、DNAは延々と伸びていくという形になっているわけです(正確には、5'のOHはリン酸基・Pになっており、そのPと3'のOHが結合します)。
話があちこち飛んで恐縮ですが、ジデオキシヌクレオチドでは、この3'のOHをなくしてやった物質なのです、ということだったのでした(なお、デオキシヌクレオチドは、2'のOHがなくなっていることが、図を見ればお分かりいただけると思います)。
するとどうでしょう、当たり前ですが、DNAは5'のPと3'のOHが結合して伸びていくということですから、このジデオキシヌクレオチドがつながったDNAは、そこから先、新しいヌクレオチドと手をつないで伸びていくことができなくなってしまうんですね!
この、「DNAの合成が途中で止まる」というのがサンガー法の最大のポイントなのです。
これだけでは全くもって意味不明かと思われますが、具体的なシーケンス反応の仕組みに踏み込みますと…
・読みたいDNAの鎖のコピーを作る(合成する)
→このDNA合成に、ジデオキシヌクレオチドを混ぜることで、途中で合成が止まる鎖が爆誕する
→DNAは、長さに応じて電気泳動で分けることが可能なので、どこで止まったのかを見れば、(1塩基ずつの長さの違いで)配列を知ることが可能!
…という流れになるわけです……が、この文章だけで理解することなどおよそ不可能なので、図を使った説明をしていきましょう。
…とその前に、もう1つ必要な知識として、「DNAのコピーを作る」などと書きましたが、これに使うのはDNA合成酵素(DNAポリメラーゼとも呼ばれますが、名前はどうでもいいでしょう)なんですけど、この酵素は、「最初の十数塩基だけ相手の鎖に結合したDNAを、5'→3'方向に伸ばす」という能力をもっています。
(逆にいうと、DNA合成酵素は、「DNAを5'→3'方向に伸ばせる(相手の鎖をコピーできる)が、その反応を進めるために、『取っ掛かり』となる短いDNAが端っこに必要」ということ。)
この十数塩基の短いDNAを「プライマー」と呼んでおり、習いたての頃は「プライマー」とか「プロモーター」とかややこし過ぎる!と憤りを覚えるのですが、まぁ名前はどうでもいいとはいえ、これはそう呼ばれている分子であり、「短いDNA」というのも逆にややこしさに拍車をかけるだけなので、鬱陶しいですがその名を使わせてもらいましょう。
つまり、「配列を読みたいDNAに、プライマーを混ぜて、DNA合成酵素で合成することで、コピーDNAを合成(伸長)する」というテクニックに、上手いことジデオキシヌクレオチドを絡めることで、配列を読むことが可能になるのです!
いやぁ~分かり辛い!
しかし、図でみれば一発かと思うので、図で説明してみましょう。
まず、手元に自分が読みたい二本鎖DNAがあるわけですね。

二本鎖DNAは、相棒の塩基と手をつないでいるとはいえ、それは弱い力でつながっているに過ぎないため、温度を上げると手が離れて一本鎖ずつに分離します(当然、温度を冷やせばまた元の二本鎖に戻ります。しかし、高温状態では、安定して手をつなげず、分かれるものが多発するということですね)。
いきなりの新知識で、何だか都合よすぎる話かもしれませんが、DNAにはそういう性質があるのでそうなのかと納得してください。
で、ここに、過剰量のプライマーを入れてやれば、一本鎖に分離したあと改めて温度を下げることで、プライマーが結合したDNAが沢山できあがることになります。

プライマーは当然、自分で設計したものであり、自分の読みたい配列の少し上流の部分の相棒の鎖に結合する配列をもったものになります(※ここが初めて聞いた時に「はぁ?」と突っかかるポイントかと思うのですが、後で改めて注で補足する予定です)。
この状態のDNA(部分的に二本鎖のDNA)に、DNA合成酵素と、DNAの構成分子であるデオキシヌクレオチド(dA, dC, dG, dT。合成の材料として使われる)を加えると…

相棒の鎖の塩基に応じたデオキシヌクレオチドが順番に取り込まれることで、1塩基ずつ、DNAが伸長していきます。
何もなければこの合成は、相棒の鎖の端っこ(図でいう右端)まで進むわけですが、ここにジデオキシヌクレオチド(ddA, ddC, ddG, ddT)を加えてやると…

ジデオキシを取り込んだ時点で、そのDNAはもう伸びることが不可能になり、そこで合成がストップするんですね。
つまり、例えばプライマーが結合した次の部分からの配列がATGGC…であったとすると、プライマーが15塩基だったとして…
[プライマー](ddA)(16塩基)
[プライマー]A(ddT)(17塩基)
[プライマー]AT(ddG)(18塩基)
[プライマー]ATG(ddG)(19塩基)
[プライマー]ATGG(ddC)(20塩基)
(以下端っこまで続く…)
…という、1塩基ずつ長さの違うDNAが、沢山生まれるわけです。
なお、ここもパッと見「は?」と思われる点かもしれませんが、この反応液の中には、デオキシ(普通に伸長が続く)とジデオキシ(伸長ストップ)の2つの合成材料を混ぜているので、どちらが取り込まれるかは、純粋に運・確率となっています。
例えばジデオキシが過剰量あったら、ほとんど全て最初の塩基にddAが取り込まれてDNAが全然伸びていかない感じで終わってしまいますし、逆にジデオキシが少なすぎると、常にデオキシが取り込まれてしまい途中で一切止まることなく反応が端まで進んでしまう…という感じになってしまうわけですが、ちょうどいい絶妙な濃度でそれぞれを加えてやることで、「止まるものもあれば、進むものもある」という状況を作り出せるわけですね。
…と、ここまで来れば、勘のいい方ですと、もしかしたらどうすればDNAの配列を読めるか思い浮かぶかもしれません。
というか上で少しもう書いていましたが、電気泳動で長さに応じてDNAを分けて、あとは「この長さのものはAで伸長が止まった」ということさえ分かれば、それで配列解読完了ですね。
そうするにはどうすればいいのか…?!
「分かりやすく説明」と書いたので、一発でバシッと全部説明を終えたかったんですけど、あまりに記事が長すぎると「うへぇ」となって分かりにくくなる気もしたので(もう既に相当「うへぇ」レベルが高いともいえますし)、中途半端ですが次回へ続く、とさせていただくと致しましょう。
(まだ全然分かりやすくないですけど、次回、全ての謎が明らかに!続きはこちら)