アミノ酸の出現頻度についてのお話、前回は散布図グラフを見て完全途中状態で終わっていました。
早速続きに参りましょう。
まずそのグラフの再掲からですが、情報を見かけたのがNIMBioSというテネシー大学所在の国立数学・生物学総合研究所の記事で……
…記事中の記述はやや説明不足だったので、頻度のグラフ画像自体は元ネタである1969年のScience誌から直接お借りしていた感じですね。
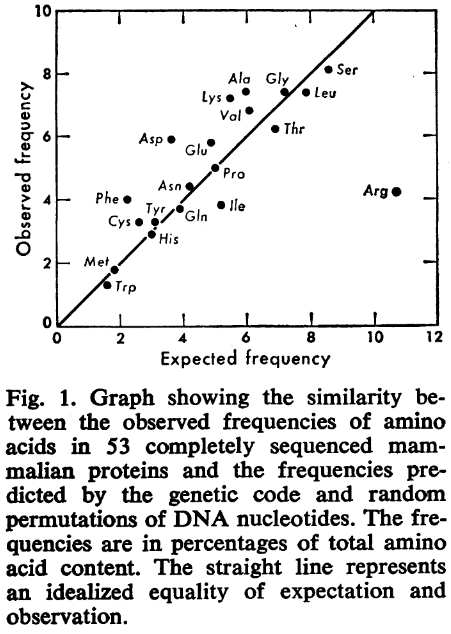
こちら、縦軸が「実際のアミノ酸の出現頻度」であり、横軸は「理論上の想定出現頻度」になるということですけど、この場合の「理論値」ってのは対応しているコドン(=取り込むアミノ酸を指定するDNAの3文字暗号)の数と、さらにDNAの各文字の存在比まで考慮したものから得られたもので……
アミノ酸に応じて、対応コドンの数は1, 2, 4, 6種類があるわけですけど、当然、対応コドンが1つしかないものは理論的に出現しにくい一方、6種類もコドンを持ってるやつは理論上じゃんじゃん登場するに決まっている…という話であり、パッと見で分かる通り、アルギニン(Arg)が、こいつは対応コドン6つ持ちのアミノ酸なわけですけど、それにしては実際の出現頻度が妙に低いことになっている……
…という所まで書いた時点で、前回は時間切れとなっていました。
果たしてこのArgの「妙な理論値とのズレ」は何を意味しているのか、ということですが、実はこの論文が主張しているのはそっちではなく、
「(Arg以外は)『理論値=対応コドンの数』と『実際のアミノ酸出現頻度』が非常に高い相関関係を誇る」
…ということでして、(改めてあくまでArg以外は)コドン1つ組・2つ組・4つ組・6つ組のアミノ酸登場頻度が、概ねグラフ内に引っ張られた直線付近に位置しているということから、
「タンパク質を構成するアミノ酸の出現頻度は、ダーウィンの唱える自然選択ではなく、DNAのコドンに大きな影響を受けていることがよく分かる」
…というのがポイントになっている形なんですね。
要は、進化論では「自然淘汰によって生物は環境に適応していく」というのが現在でも主流の説であり、生物種のレベルでは概ね正しいわけですけど、もっとミクロなタンパク質レベルだとどうも必ずしもそうではないらしい…
(なぜなら、もし自然淘汰によってのみタンパク質を構成するアミノ酸が決まっていくなら、各アミノ酸を指定するコドンの数に影響など受けるはずがないので。
実際はグラフにある通り、概ねアミノ酸の出現頻度は対応コドンの数に強く相関したになっていますから、「タンパク質を構成するアミノ酸の頻度は、確実にDNAコドンの影響を受けている」って話ですね)
…ということを主張しているのがこの論文なわけですけど、まぁその辺の分子進化生物学については小難しい話になりますし、聞いても「だから?」としか思えない感じなので省略するとしましょう。
「ポイントではない」としたものの、当然Argだけが例外的に少ない(といっても、「理論値より少ない」であり、実際の出現頻度的には、ちょうど真ん中ぐらいに位置している(=縦軸のみでの評価)わけですけどね)のは興味深い話で、この論文でもディスカッションされていたわけですが、著者のキングさんとジュークスさんは、
「脊椎動物のDNAでは、CpGという配列が非常にレアであることが知られており、それがArgの登場頻度が妙に低くなった理由であろう」
…という考察をされていました。
CpGというのは、普通にDNAの塩基が「CG」の2連続で続いていることを表しているだけなのですが…
(DNAの塩基は、リン酸基=「p」を介してつながっていますが、あえてそのpを明記した形ですね。
その辺の話は、ずーっと前のこの辺の記事で、ちらっと触れたことがありました↓
…普段は省略する「間のp」をなぜ明記するかというと、DNAが二本鎖を形成する際のペアは「A⇔T、C⇔G」だったわけですけど、「CG」という記述だけでは、その「二本鎖のCGペア」と紛らわしいから、ってのがその理由だといえましょう)
…とまぁ表記法についてはともかく、実は「CG」の並びというのは若干レアである…というか特別な意味を持っていることが知られていまして、具体的には「CpGアイランド」などと呼ばれるように(参考:Wikipedia記事(↓))……
…これは、しばしば遺伝子のプロモーター領域にまとまって存在して、遺伝子のON/OFFの制御をしていることなんかで知られているわけですが、そういった特別な使われ方がされていることもあり、遺伝子のタンパク質を指定するコード領域にはあまり出てこない形になっているのかもしれませんね。
実際の論文においては、1969年時点では恐らくまだCpGアイランドの存在も知られていなかったのでそこには言及されておらず、他の進化的な考察が色々となされていましたがまぁ今回はあまり関係ないのでその辺もともかく……
Argのコドンは「CGU, CGA, CGC, CGG, AGA, AGG」と、6つ中4つも「CGn」の形になっているので、「理論上はコドンが6つもあり、しかも存在比の多いAとGに富む」ことから一番出てきて然るべきなアミノ酸かと思いきや、実は哺乳類の遺伝子DNAには「CG」2連続が異様に少ないという別のポイントもあり(当時のデータで、「理論値の10%未満しか存在しない」と書かれていました)、結果、Argは「コドンの存在比だけから見た理論上の頻度」よりもぐっと低い実際の登場頻度になったのだろう、と、そういう形でまとめられていた感じです。
なお、引用記事にも論文にも、先ほど貼ったグラフ以外に具体的な数字を伴った表も紹介されていたので、こちらも引用させていただきましょう。
コドン
脊椎動物における、実際の出現頻度
Alanine
アラニン
7.4 %
Arginine
アルギニン
CGU, CGA, CGC,CGG, AGA, AGG
4.2 %
Asparagine
AAU, AAC
4.4 %
Aspartic Acid
GAU, GAC
5.9 %
Cysteine
UGU, UGC
3.3 %
Glutamic Acid
GAA, GAG
5.8 %
Glutamine
グルタミン
CAA, CAG
3.7 %
Glycine
GGU, GGA, GGC, GGG
7.4 %
Histidine
CAU, CAC
2.9 %
Isoleucine
イソロイシン
AUU, AUA, AUC
3.8 %
Leucine
ロイシン
CUU, CUA, CUC, CUG, UUA, UUG
7.6 %
Lysine
リシン
AAA, AAG
7.2 %
Methionine
AUG
1.8 %
Phenylalanine
UUU, UUC
4.0 %
Proline
CCU, CCA, CCC, CCG
5.0 %
Serine
セリン
8.1 %
Threonine
ACU, ACA, ACC, ACG
6.2 %
Tryptophan
UGG
1.3 %
Tyrosine
UAU, UAC
3.3 %
Valine
バリン
GUU, GUA, GUC, GUG
6.8 %
Stop Codons
停止コドン
UAA, UAG, UGA
---
…これをグラフにしたのが例の図だったわけですが(出現頻度を合計したら、当然100%ですね)、「理論値とのズレ」とか進化論的にうんぬんはよぉ考えたらどうでもいいので、この表にもある実際の出現頻度のみに着目してみますと(まぁ上下関係は表よりグラフの方が分かりやすいですけどね)……
最多出現頻度のアミノ酸は、セリン→アラニン→ロイシン→グリシン→…という順番になってるわけですが、まぁセリンは「-OH基」があるものの、こないだこれも「大して特徴のない雑魚アミノ酸」などと書いていた通り、やっぱりタンパク質のほとんどは、基本的にそこまで大きな特徴のないつまんないやつらが数合わせ的に大量に存在して形を作り、より特徴あるやつがたまにピンポイントで存在することで、特別な機能を生み出していることが多いんだね……なんてことがいえましょう。
ちなみに5番目はリシンでしたが、これは正直意外ですね。
リシンは塩基性アミノ酸で、正直、電荷(プラスマイナスチャージ=酸性アルカリ性ですね)もなければ極性もない他の雑魚アミノ酸の方が普通にもっとありそうな印象だったんですけど、少なくともこのデータからはリシンがNo. 5だということでした。
というかそもそも「このデータは何のタンパク質を調べたものなの?」というのがまず気になる点で、それについては論文のオリジナル画像の説明文まで貼っていたのに触れていませんでしたが、これは、その説明文にあった通り、「1969年当時で既に配列が完全に解明されていた、53個の哺乳類タンパク質」に基づくデータであるとのことです。
人間には2万を超える遺伝子が存在すると言われていますが、1969年なんてまだヒトゲノムが解明されていなかったどころか、PCRすら発明されておらず、偉大なるサンガーさんが現在でも用いられている素晴らしいDNA配列解読法・サンガー法を開発するのもこれより待つこと数年が必要……という時代だったわけで、流石にヒトの持つタンパク質全部を解析して出されたデータではない感じなんですね。
(ちなみにサンガー法については、この辺の記事(↓)から何回かかけて見ていました。)
いずれにせよ、僕の予想としてはやはり例のBCAA(バリン・ロイシン・イソロイシン)なんかが最も多く含まれるアミノ酸である印象があったんですが(改めて、化学的には何の特徴もないからこそ、タンパク質全体の基本構造を作るのに打ってつけで、だからこそサプリとかで大量に摂取することがオススメされてるわけですね)、特にロイシンは無駄に対応コドン6つ持ちであることからも、多くのタンパク質でメインとなって使われるアミノ酸である印象があったわけですけど、ハナ差でセリンの方が多いという感じでしたか。
まぁ、セリンもコドン6つ持ちですし、非常によく使われるアミノ酸である印象がありますけどね。
一方頻度ワースト5は、少ない方から、最デカのアミノ酸であるトリプトファン、開始コドンであるメチオニン、塩基性かつリングもちのヒスチジン、硫黄もちでタンパク質の構造に大きく影響を与えるシステイン、リング&OHもちのチロシン……となっているようで、やはり特徴のあるものやデカブツは、あまり顔を出さない感じといえますね。
(もちろんメチオニンは開始コドン以外にも、一般のコドンとしても普通に登場しますが、まぁこいつも硫黄もちですしね、対応コドンが1つであることからも、Metは(開始コドンとして必ず存在しているにもかかわらず)結構レアなアミノ酸の印象です。)
ちなみに、「CGn」というコドンは少ないという話でしたが、アミノ酸以外にも、コドンの頻度も普通に、ゲノム時代の今では簡単に解析されている感じです。
そんなわけで実際どのぐらいの頻度になっているのか、「codon frequency」で検索してみたら、僕もお世話になったことがあるバイオ試薬・解析系の企業Genscript社が、ヒトのみならず色々な生物種のコドン頻度を表で紹介してくれていました。
(表がメインのページだからか、リンクカードは取得されませんでしたが、こちらですね↓)
https://www.genscript.com/tools/codon-frequency-table
色々な生物のコドン頻度表がまとめられていますが、今回はやはりヒトのコドン頻度テーブルを、こちらも項目名を翻訳して引用させていただきましょう。
コドン アミノ酸 割合 頻度(千分率) 数 コドン アミノ酸 割合 頻度(千分率) 数 TTT
TTC
TTA
TTGF
F
L
L0.45
0.55
0.07
0.1316.9
20.4
7.2
12.6336562
406571
143715
249879TCT
TCC
TCA
TCGS
S
S
S0.18
0.22
0.15
0.0614.6
17.4
11.7
4.5291040
346943
233110
89429TAT
TAC
TAA
TAGY
Y
*
*0.43
0.57
0.28
0.2012.0
15.6
0.7
0.5239268
310695
14322
10915TGT
TGC
TGA
TGGC
C
*
W0.45
0.55
0.52
1.009.9
12.2
1.3
12.8197293
243685
25383
255512CTT
CTC
CTA
CTGL
L
L
L0.13
0.20
0.07
0.4112.8
19.4
6.9
40.3253795
386182
138154
800774CCT
CCC
CCA
CCGP
P
P
P0.28
0.33
0.27
0.1117.3
20.0
16.7
7.0343793
397790
331944
139414CAT
CAC
CAA
CAGH
H
Q
Q0.41
0.59
0.25
0.7510.4
14.9
11.8
34.6207826
297048
234785
688316CGT
CGC
CGA
CGGR
R
R
R0.08
0.19
0.11
0.214.7
10.9
6.3
11.993458
217130
126113
235938ATT
ATC
ATA
ATGI
I
I
M0.36
0.48
0.16
1.0015.7
21.4
7.1
22.3313225
426570
140652
443795ACT
ACC
ACA
ACGT
T
T
T0.24
0.36
0.28
0.1212.8
19.2
14.8
6.2255582
382050
294223
123533AAT
AAC
AAA
AAGN
N
K
K0.46
0.54
0.42
0.5816.7
19.5
24.0
32.9331714
387148
476554
654280AGT
AGC
AGA
AGGS
S
R
R0.15
0.24
0.20
0.2011.9
19.4
11.5
11.4237404
385113
228151
227281GTT
GTC
GTA
GTGV
V
V
V0.18
0.24
0.11
0.4710.9
14.6
7.0
28.9216818
290874
139156
575438GCT
GCC
GCA
GCGA
A
A
A0.26
0.40
0.23
0.1118.6
28.5
16.0
7.6370873
567930
317338
150708GAT
GAC
GAA
GAGD
D
E
E0.46
0.54
0.42
0.5822.3
26.0
29.0
40.8443369
517579
577846
810842GGT
GGC
GGA
GGGG
G
G
G0.16
0.34
0.25
0.2510.8
22.8
16.3
16.4215544
453917
325243
326879
まぁコドン表ではなく頻度表なのでかもしれませんが、非常に珍しく例の4×4×4フォーマット以外のパターンでなんとなく見づらいですけど、まぁ形はともかく……
これを見てみると、やはりアルギニンRのCGnは、特にCGTやCGAなんかは顕著に存在比率が低いといえますね!
(さらに、TCG, CCG, ACG, GCGなんかも軒並み一桁割合(「一桁パーセント」と言いたくなりますが、千分率なので、千=ミレニアムの「mille」ですから「一桁パーミル」って感じですかね)で、かなり低いことが窺えます。
こんな感じで、科学技術の発展により今ではコドンの頻度まで分かっているわけですが……って、よぉ考えたら、1969年の古臭ぇデータなんて見なくても、これの各コドンの千分率頻度を合計したら、各アミノ酸のより正確な出現頻度になるやん!
…と唐突に気がついたので、サクッとエクセルで作成してみました。
(最初っからこれを見ればよかったですね(笑))
ズバリこれが、より正確な、ヒトゲノム情報をもとに計算された、ヒトのタンパク質におけるアミノ酸の出現頻度だ!

お~、やっぱりロイシンがナンバーワン!
(千分率なので、「1000分の○」ですから、ロイシンは実に全タンパク質の1/10程度(=99.2/1000)も占めるということですね。)
何気にグルタミン酸が躍進してきたり、BCAA様の他の2名ValとIleは結構下位に沈んでいるなど(そしてリシンもやっぱりそこまで上位じゃなかったですが、でもその代わりに酸性アミノ酸であるグルタミン酸がかなり多い、というのは意外です)、面白い結果でしたが、こんなグラフをちまちま作っていたら全く時間がなくなってしまいました。
もう概ね見たかった情報は見終えたわけですけど、本来の脱線元のネタに戻ってみるなど、触れてみたかった点はもう少し次回へ続くという感じにさせていただきましょう。