前回の記事ではかなり網羅的で分かりやすいtRNAデータベースを参照することで、tRNAの配列の違い(同じアンチコドンをもつtRNAは完全に一致しているのか?⇒ほとんど同じだけど、一部微妙に違うのもあり)を見て楽しんでいました。
このデータベース、何気に用意されている情報が豊富で案外面白く、ついついその後ちょっとの間見耽ってしまいましたね。
色々見てたら触れておくのもいいかなと思えた話があったので、今回はまた脱線でちょっとtRNAの配列を垣間見てみるといたしましょう。
まず、前回は酵母のtRNAを見ていましたが、このデータベースにはヒトtRNAの情報も用意されていました。
ヒトのtRNAの配列についてじっくり見たことがなかったので、この機会にどんな具合かちょっくら覗いてみるのも良さそうです。
以前ゲノムブラウザーの記事でも触れたことがありましたが、ヒトゲノム情報の最新版はGRCh38と呼ばれるデータセットなんですけど、このサイトではGRCh37が「最もよく見られているゲノム」のトップに来ていたので、まぁtRNA解析では一代前の37の方がよく使われているのかもしれないねということで、こちらにアクセスしてみましょうか。
クリックすると、例の、tRNAの遺伝子と配列の違いを整列して一覧表示してくれたものがズラーッと、アミノ酸のアルファベット順に、アラニンtRNAから順に列挙されています。
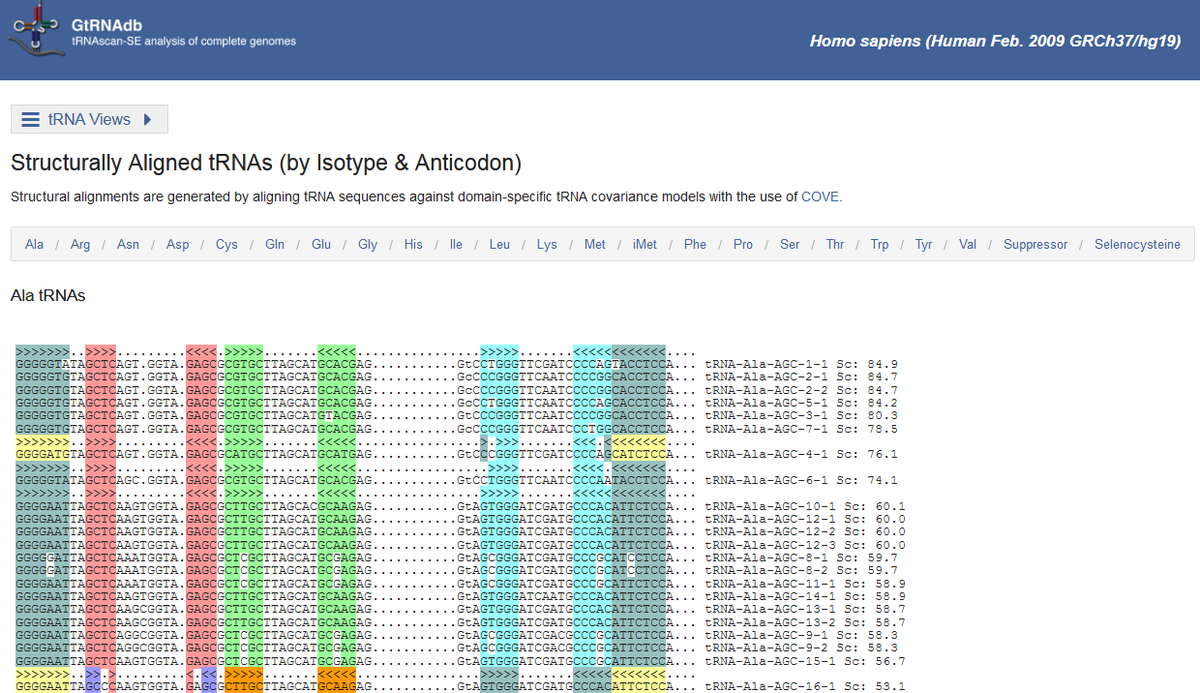
ちなみにこういう配列比較をアラインメントといいますが、このサイトでは、二重鎖(ステム)を組むことが同じ色でハイライトされることで明示されており、どこがステムを組んでいるのかそれなりに分かりやすい感じになっていますね。
(例えば、暗い青色が最初と最後にありますが、この7塩基ずつがステムを組むわけですね。tRNAのよくある二次構造図を思い浮かべていただくと、確かに両末端はステムになっていました。
ちなみに、色などなくても、実は「>」と「<」の記号のみでステム形成は表示可能です。
tRNAはシュードノット(シュードノットは、この記事で触れていました)を形成しないので、「<」記号は必ず直近の「>」とペアを形成することから、>と<だけで一意に定まっているという形ですね。
→例:一番上に不等号記号も表示されていますが、最初の暗い青が「>」記号、次の赤も「>」記号で、その次に「<」記号が出てくるけど、これは直近の「>」記号、つまり赤い部分とペアを組む(=一個飛ばして暗い青の部分と組むことはない。そういう形で組んでしまうのがシュードノット)ので、まさに色で表示されている通り、初出の「<」は直近の「>」とステムを組むということ。)
まぁアラインメントの見方はともかく、これを見てみると、アラニンを運ぶ用のAGCアンチコドンをもつtRNAは、まさかの、tRNA-Ala-AGC-1-1から16-1まで、16種類も存在!
完全に同一だったら2-1と2-2のように、同じ番号が振られるので、この16種類は、全て微っ妙~に異なるものになってるということですね。
そう、前回は「同じアンチコドンをもつtRNAなら、基本同じ配列だと思います」とか抜かしていましたが、ヒトのtRNAを見てみると、全然そんなことにはなっていませんでした(笑)。
まぁ、それぞれの違いは1塩基ずつぐらいなので、「ほとんど同じ」とはいえるものの、1塩基でも違うといえば違うので、「基本同じ」は全然正しくなかったですね。
どうも適当言ってすみませんでした、お詫びして訂正しておこうと思います。
やはりヒトは酵母に比べて圧倒的に高等で複雑な生物なので、微妙に違う色々なバリエーションのtRNAをもってるといえるのかもしれませんね。
その点に関してもまた後ほど別のご質問と絡めて触れようと思いますが、せっかくなのでヒトtRNAには本当に色々なバリエーションがあるのか、もしかしたらアラニンtRNAだけなのでは…?と思い、順番に下まで見てみましたが、まぁどのtRNAも見事にバリエーション豊かなことで…。
最もバリエーションの少ないものは、ヒスチジン(His)を運ぶtRNAで、これでも2種類の異なる配列があるみたいですね(1種類の配列しか存在しないtRNA分子は、軽く見た限りありませんでした)。

とはいえ、こちらは9つが全く同一(別の染色体にあるけど、完全に同一配列)で、異なる配列の遺伝子はたった1つ(2-1)しかないので、前回の酵母のtRNA-Ser(AGA)に近いものがありますね。
せっかくなので関連して、前回は「異なる生物なら、同じアンチコドンでも配列は異なるでしょう。ただ、近い生物種なら、似てることもあるかもね」とか書いていたので、そちらも確認してみましょうか。
何と比べようかと思いましたが、ヒトに近い生物はヒトほどゲノム解析が進んでいない気がするので、今回はよく研究されているモデル生物で、酵母みたいな単細胞カスではなく、せめて哺乳類の、マウスなんかと比べてみよう、と思い立った次第です。
ちなみに、マウスの学名はMus musculusで(ヒトの学名がHomo sapiensであるのと同じ感じで、英語のMouseとも違う、学名がある感じですね)、最初の画像の「よく検索されているゲノム」にも挙がっていました。
なお、マウスというのはハツカネズミのことで、小さめの、まあまあ可愛らしいタイプのネズミさんですね。
研究に用いられるのはマウスが多いです。
ちなみに、汚い都会で見かけることのあるのはラット、いわゆるドブネズミで、より大きくてグロい、ドブカスみたいなクズですね。
とりあえず今回はラットではなくマウスのtRNAを見ていくとしましょう。
とはいえまぁ、今調べたいのは配列の比較なので、ここは改めてBLASTを使う場面でしょう。
検索対象生物種をマウスにして、ヒトのtRNA配列をBLASTにかける感じですね。
まずは、一番バリエーションが少なく、ほとんどが同じ配列として存在していた、tRNA-Hisから調べてみますか。
BLAST検索の結果が、こちら!
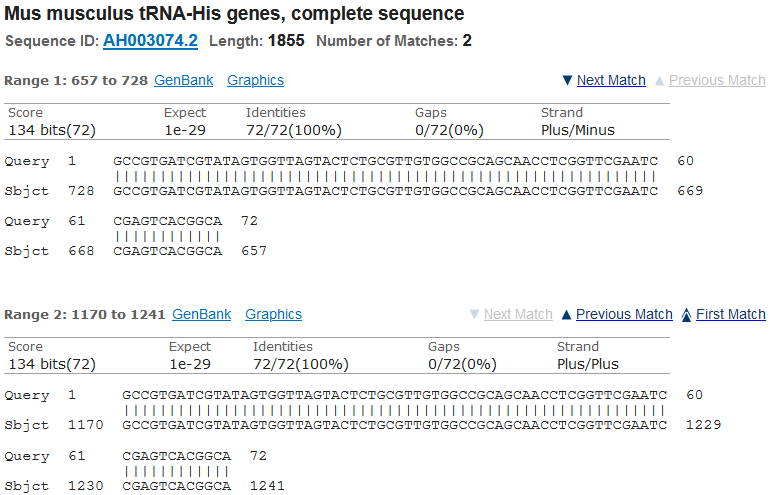
ぬわんと!
ヒトのtRNA-HisとマウスのtRNA-Hisは、72塩基、100%完全に配列が一致!!
まさか!誇り高き全生命体の頂点に立つ我々人間様と、寿命が2年ぐらい、野生では捕食者が多いので平均4ヶ月程度しか寿命がないといわれる、ネズカスごときが全く同じものを使っているだなんて!!
これはヒトとしての尊厳が踏みにじられるかのごとき由々しき事態でカチンときました、たまたまこのtRNAが同じだっただけで、きっと我々知恵ある生命体は、基本的にはネズ公さんなんかとは違うものをもっていることでしょう。
先ほど見ていた、バラエティーに富んでいるtRNA-Alaなら、きっと害獣マウカスとは違う、高級なtRNA(何だよソレ(笑))をもっているに違いありません。
そんなわけでtRNA-Ala-AGC-1を使ったBLAST検索、あらよっと!
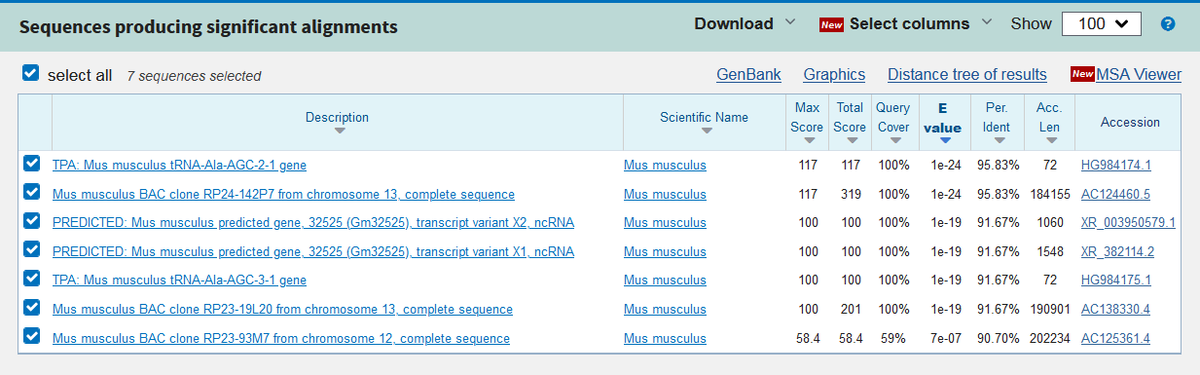
ね?
マウスとの一致率は、95.83%!
詳しく見てみると…

3塩基も違いますねぇ!
…って、害獣とわずか3塩基しか違わないのは、それでいいのか(笑)、って気もしますが、その3塩基がヒトとケダモノを分ける重要なポイントなのです。
ま、一応他のtRNA-Alaも見ておきますか。
多分人間様とネズチューごときとは違うと思いますけど?

…ってグアァーー!!
結局、こちらはマウスtRNA-Ala-AGC-3の配列と、完全一致!
人間はネズミと同然だった…?!
そんなバカな、我々はホモサピエンス様だぞ…?と地球上の頂点に立つ生物の奢り昂ぶりを隠し切れずに震える手で次のtRNA-Argを検索してみるも…
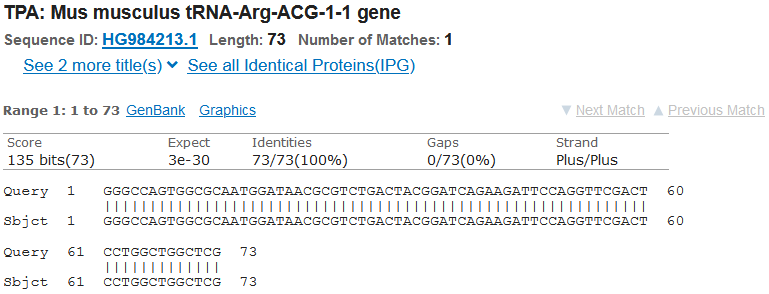
ギョエェェー!!
またしても100%一致!!
おかしい……こんなことは許されない…と続くtRNA-Asnは…
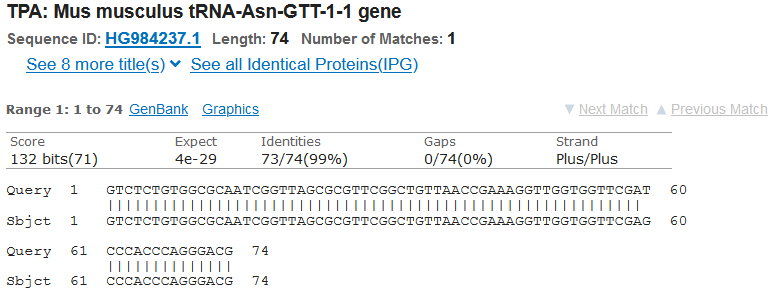
っしゃオラァーー!!
1塩基違い!!
やっぱりクソザコネズゴミなんてこんなもんよ!
…と思いきや、その次にあったtRNA-Asn-GTT-2も一応BLAST検索してみると…

ギャヒョーーン!!
結局またしても完全一致のものも存在!!!
ぐぬぬ、人間も所詮ケダモノということか…と、プライドをズタズタにされながら検索を続けるも、続くtRNA-Asp, -Cys, -Glu全てがいきなり100%一致!!
まさか、我ら人間様は全てのtRNAでマウスと同じものを使っている…??と絶望の淵に突き落とされかけたその刹那!!
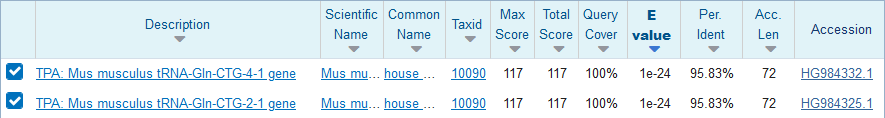
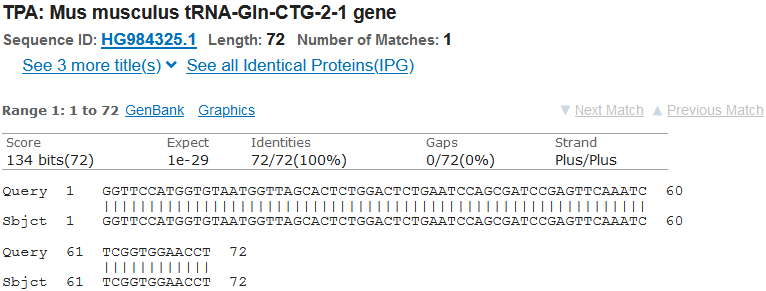
グルタミンのtRNA、これは一致度が低い!
…といっても先ほどと同じ3塩基違いか…。
しかし、ヒトtRNA-Glnは他のも割と似てる配列だからね、これは、「マウスなんぞとは違うtRNAを使っている」パターン発見の大チャンス?
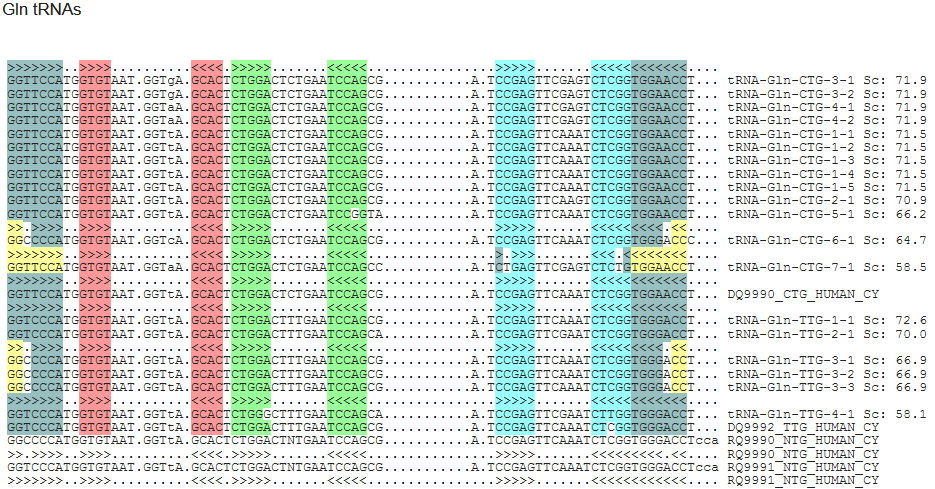
…と思ったら、その下にあるtRNA-Gln-CTG-1でいきなり100%キチャッター!
…という似たようなことをグダグダ上から順番に繰り返すも、どのアミノ酸のヒトtRNAも、すぐ100%一致マウスtRNAが見つかる始末…。
と、ここでコドンが6種もあることでおなじみ、Leu(ロイシン)の出番だ!
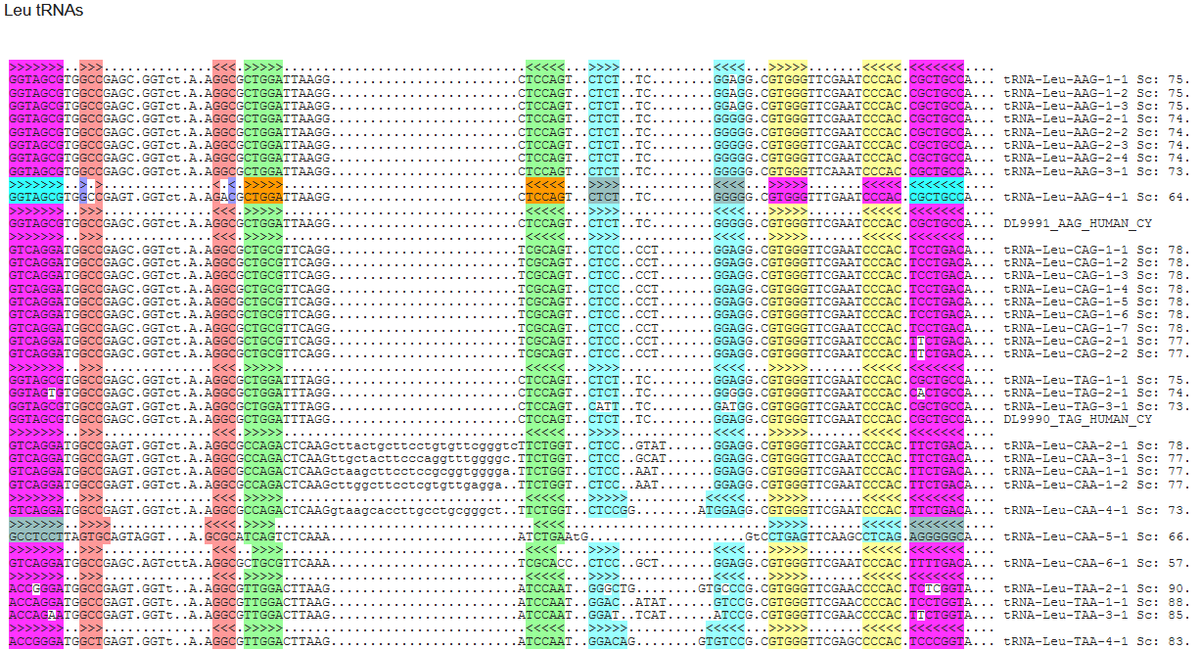
ロイシン先生、頼みますよ…
まず最初のtRNA-Leu-AAG-1は1塩基違いでGood!!

しかも違いはアンチコドン部、つまり同じアンチコドンのものは結構違う(相同性スコアが低い)から別のtRNAがヒットしてきたという推測が成り立つわけで、これは、もしや来るか?と思いきや…
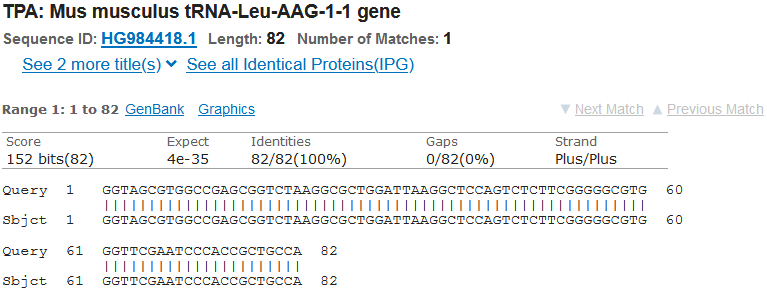
…と、したり顔のうんちくとともに披露したしょうもない期待も即撃沈のあっさり一致、ヒトとマウスで違うやつ、全然こねぇー!!
まぁ、実は各アミノ酸のトップにあるやつ(最も遺伝子の数が多いやつ)を見ていただけなので、遺伝子のバリエーションが少ないやつ(下のほうに列挙されているやつら)なら、今までの中にもヒトとマウスで完全一致しないやつもあったかもしれませんけどね。
とりあえずまずは、各アミノ酸用tRNAのトップにあるやつ(最も異なる種類の配列をもつもの)だけ比較していくとしましょう。
それが男ってもんです(こんなのしょうもない話で、何がだよ(笑))。
…と、続いては…あっ!メチオニン!!
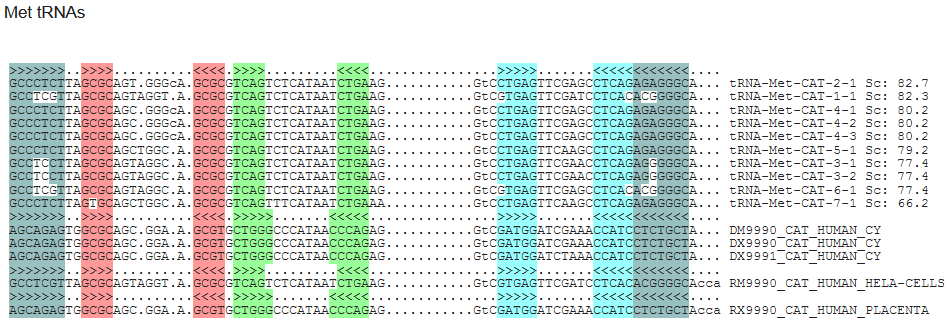
こいつは、開始コドンになったりと重要なアミノ酸だからね、きっとヒトとマウスとで違う配列に違いない!!
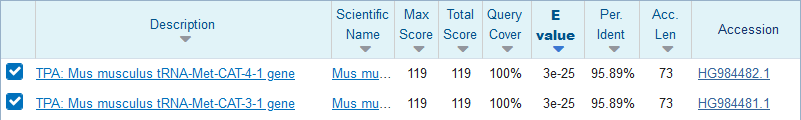
一発目、ほら、一致率95.89%、かなり違う!
しかもtRNA-Metは数が少ない、チャンスだ!!(と思いきや、コドンは1種類しかないけど、tRNAは7種類もあるようなので、結構多いですね)
さぁ来い、続くtRNA-Met-CAT-1との比較は…!
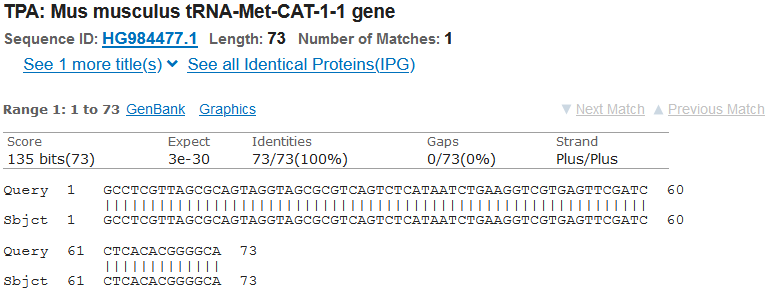
ギョエーン、あっさり一致!!
…ま、そもそも、tRNA-Metは、開始コドンで使われるものはtRNA-iMetと、別のものがあるんですけどね。

…ってまぁ、そのtRNA-iMet-CAT-1も、一発で完全一致KOでしたが…。
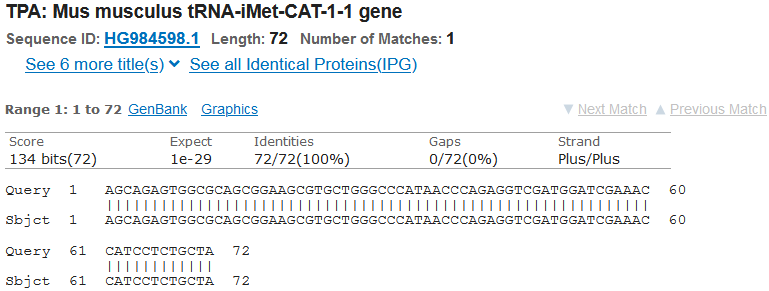
…と、なおもガンガン見進めていくも全てあっさり100%一致、そろそろtRNAがなくなってきた…。
残る希望の星は、同じくコドンが1種類しかないTrp、これは、何となく僕のカンピューター的に、めちゃくちゃ期待大に感じるわけですが、果たして…??
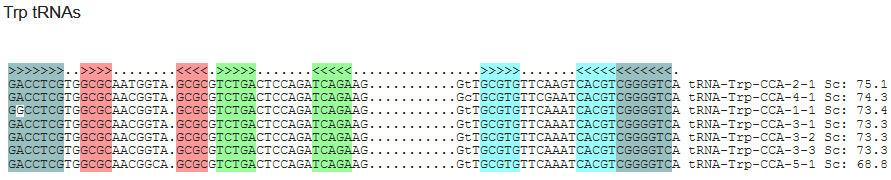
結果、いきなり完全一致、カンピューター、完全に錆び付いてたぁー!

…と、もうほとんどアミノ酸は残されていないけれど、果たして人間とネズミとで違うtRNA分子をもっている(配列が完全一致はしない)パターンは見つかるのか…?
それとも、人間もしょせん動物、チューチューマウスと全く同じものを使って生きているという、悲しき結果なのか…?!
中途半端ですが(展開も中途半端だし、そもそもの話の内容が、引っ張るほどのもんでも何でもないショボさ(笑))、冬時間でちょっと時間もなかったため、続きは次回にまわさせていただきましょう!!