そんな所で、そろそろ次のステップへ……と思いきや、何気にもう一点、第一ステップである転写について触れておいてもいいかな、って点が残っていました。
それこそが、何を隠そう、ターミネーター!!
♪デデンデンデンデデン…

…って、T2のT-1000ではなく、そういえば偶然名前だけはそっくりの、T7ターミネーターについてですね。
こないだ、lacオペレーターまで見終えたことで、「これでマップに掲載されている要素は全て把握できたことになりますね」などと書いていたんですが、 実は1点、全く触れていないものも残っていたのでした。
改めて何度も引用させてもらっているpET-15bプラスミド公式マップですが…
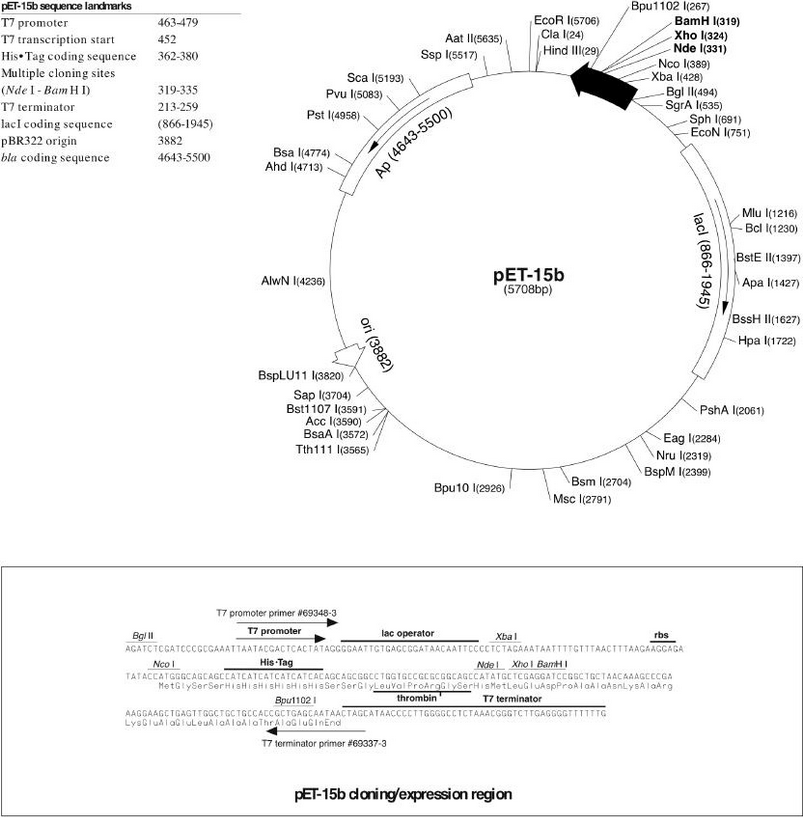
大量にある制限酵素認識部位はともかく、ほぼ全てのエレメントを一応一言程度には見ていた形ですけど、何気にT7 terminatorだけは、説明も面倒というか唐突に感じたので、完全に無視していたんですね。
しかし、転写についてサワリの部分には何度か結構つっこんで見終えましたし、ターミネーターにもごく簡単に触れておくとしましょう。
まず当たり前ですけど、ターミネーターはその名の通り、「終末をもたらす者」みたいな意味ですから、これは転写の終結に働くエレメントになります
で、転写は「RNAポリメラーゼがDNAの鋳型鎖の配列を基に、相棒となる塩基をひたすらつないでいく。DNAが続く限り、RNAの合成も続く!」……と以前の記事で自作の図なども交えて書いていたわけですが、そうすると、恐らく疑問に思った方もおられるかもしれないんですけど、「えっ?プラスミドは丸いんだから、『DNAが続く限り』って、永久にぐるぐるRNA合成が続くってこと?」って懸念が生じるわけですね。
もちろんそんな無駄にずーっとRNAが伸び続けるのは望ましくないので、必要な分が転写されたら、その時点でもう合成はストップさせたいわけです。
そこで使われているのがターミネーターで、このエレメントがあると、RNAポリメラーゼは「おっ、転写終結シグナルか。合成やめたろ」となって、RNA合成がそこで終わってRNAポリメラーゼは鋳型として読んでいたDNAから離れる形になります。
これも大学の生命科学の最初ぐらいに習う話ですが(高校生物ではほぼ触れないはず)、単純な仕組みなので学ぶ取っ掛かりに打ってつけの大腸菌(細菌類)の転写終結には、2種類の異なる仕組みが知られています。
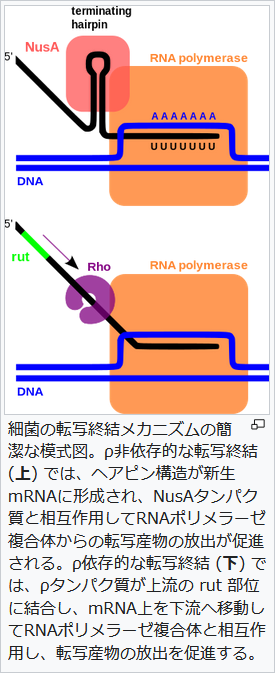
ρ(ロー)因子と呼ばれるタンパク質が関わるか関わらないかの2パターンですが、上がρなしでターミネートされるタイプで、こいつはUが数塩基連続で並ぶUストレッチという部分と、その上流にRNAがヘアピン型のステム・ループを形成することが特徴となっており、一方下がρ因子の関わるタイプで、終結部位の少し上流にrutと呼ばれるρタンパク質の結合する領域があって、ここに結合したρタンパク質(図ではRhoと表記)がスルスルとポリメラーゼとDNAとがバブル構造を形成している(DNAを読んでいる)部分まで移動し、「もう終わりだよこの転写」という感じでRNA合成を終わらせる、って仕組みですね。
ちなみに、T7ターミネーターは、(字が細かくて見づらいですが)先ほど貼った配列詳細図の最後がTTTTTTGとなっていることからも分かるように、Uストレッチとヘアピンが絡む、ρ非依存的なタイプです(改めて、DNAのマップなのでTとなってますが、RNAに転写されたらUになります)。
…と、まぁそんな感じの説明がされる話ですけど、正直、転写終結は、プロモーターの特定の塩基から確実に始まることが完璧に分かっている転写開始ステップと比べて、割と適当で、正直細部は謎というか、そこまでの厳密性もなく、よく分かってないことも多い感じです。
たとえば緑色のrut領域は、特に「これ」という決まった配列の法則は見つかっておらず、単に「Cが多め」というしょうもないルールしかないようですし、「Uが何塩基か続く」というUストレッチも、「じゃあコドンにUが続いたらどうするわけ?しかもたまたま上流がヘアピンを形成しやすい配列だったら…??」とも思えますし、案外ガバガバなのがターミネーターだ、というのが実際に思えます。
例えば、T7ターミネーターのメカニズムが調べられた昔の論文を紐解いてみたら…
pubmed.ncbi.nlm.nih.gov
割と古い(といっても1994年ですけど)、マクドナルドさん他によるものが最初にヒットしたので、名前も面白いしこれを簡単に取り上げさせていただくと…
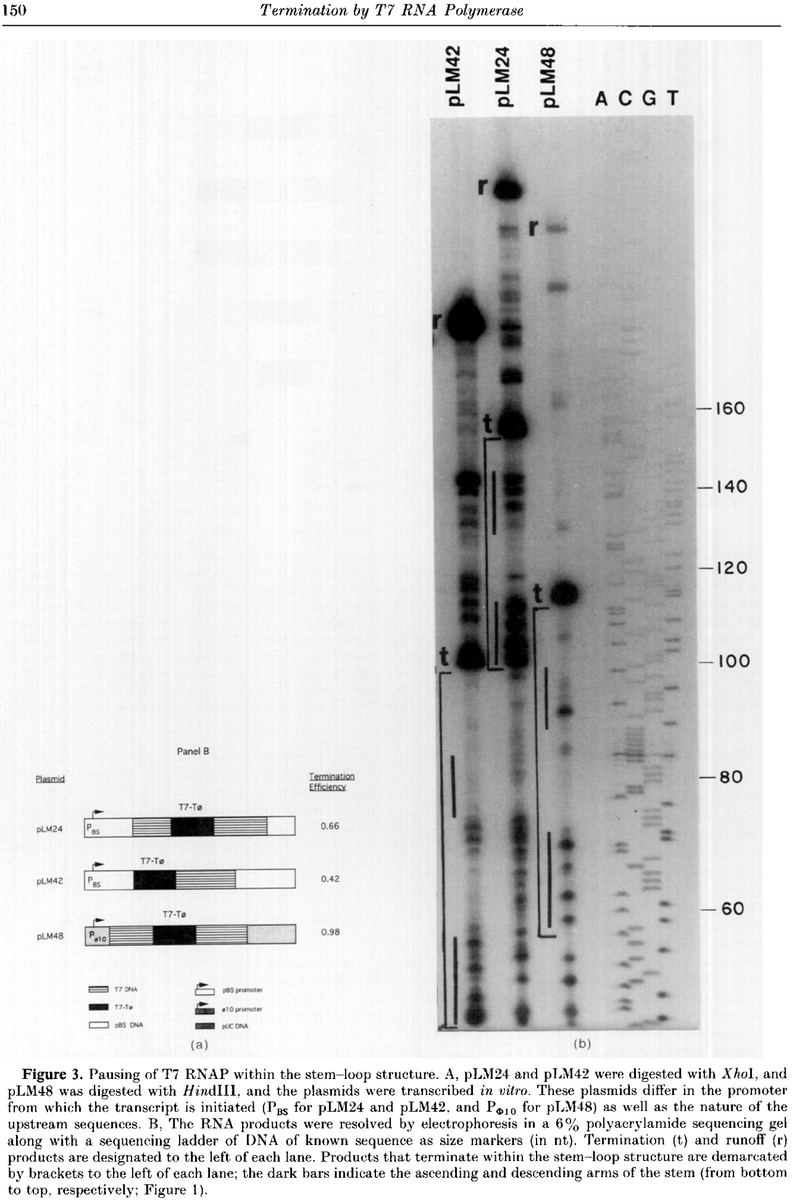
pLM42、pLM24、pLM48という三種類のプラスミド(プラスミド名は、例によってマクドナルドさんのイニシャルですね。マクナルさんが42番目に作ったプラスミド、って感じでしょう)があり、これらはターミネーターの場所をちょっと変えたり、プロモーターやターミネーター上流の配列が少し違うもののようですが、それぞれのプラスミドからの転写産物(RNA)をゲルに流して長さを解析してみた結果ですね。
図の、RNAバンドの横に示されたtが本来ターミネーターで止まる位置、一方rが、ターミネーターでは止まらずに端っこまで辿り着いた末端(プラスミドは、制限酵素で切断してあるので、端っこがあります)です。
pLM42と24はT7ターミネーターの位置を変えただけですが、それだけでターミネーターによる終結効率(ちゃんとtで止まっているRNAの割合)が結構変わっているし(=pLM24は66%がちゃんと止まってるのに、pLM42は42%しか止まらず、結構漏れ進んでいる)、もっと劇的なのが、プロモーターやDNAの中身(配列)を変えたpLM48では、この場合98%もしっかり本来のターミネーターの位置で止まっているということで、もう完全に、配列のちょっとした違いによってターミネーターがめっちゃ働くこともあればそうでないこともある、みたいな感じなわけですね。
…って、こんなのいきなり見せられても何も分からないかもしれませんが、改めて、(そもそもターミネーターより前にあるステムの部分(図で、黒いバーが横についている所がステム=二重鎖。今更ですが、ゲルの下にあるバンドほど、上流で止まっている=短いRNAですね)でも結構転写が止まっとるやん、って話含め)転写の終わりは、始まりに比べてガバガバだ、ってことですね。
(というか、T7ターミネーターの話のみならず、ずっと前この記事などで紹介していた「DNAシークエンシング」の実例があったので、「これですこれ、前言ってたのは」ってのを紹介すべくあえて触れてみた図でした(笑)。ゲルの右半分が、例の、古典的なddNTPによるシークエンシング反応ですね。この実験では、配列ではなく、ただサイズを見るためだけに使われているもののようですが。)
なお、これまたそもそも論として、プロモーターは転写において絶対必要ですが(ないとRNA合成が始まらないので)、ターミネーターは最悪、別になくても致命的な問題にはなりません。
最終的に必要なのは開始コドンから停止コドンまでの領域ですから、多少RNAが長くなったとしても、別にタンパク質が作られる上では全く関係ないんですね。
実際、先ほどの論文画像にもある通り、転写はターミネーター領域で100%止まるとは全然限りませんけど、別にそれで出来上がるタンパク質に影響はないわけです(いずれにせよタンパク質合成は停止コドンで止まるから)。
pET-15bの例でいうと、仮に転写が止まらずじわじわ進んでしまった場合でも、先ほど再掲したマップを見れば明らかなように、黒塗り矢印が進んでもちょうどアンピシリン耐性遺伝子(β-ラクタマーゼというタンパク質)が読まれるだけで、ちょっとβ-ラクタマーゼが増えても大勢には一切何も影響はありません。
ちなみに、以前、「mRNAワクチンを作る際はT7 RNAプロモーターが使われているようです」と書きましたが、この場合恐らくほぼ間違いなく、T7ターミネーターはそもそも使われていないことでしょう。
ちょうど先ほどの論文にもあったように、鋳型となるDNAが(環状ではなく)直線状で端っこさえあれば、ターミネーターなどなくともRNAポリメラーゼはそのままDNAから離れてサヨナラしますから(先ほどの論文のrは、run-offのr)、ターミネーター領域など一切不要なのです。
世界中で使われるワクチンを大量に作るためには、1塩基でも短い方が圧倒的に材料の節約になりますから、実際に使われている配列は存じ上げませんけど、まず確実にターミネーター領域なんかは省略されていると思われます。
それだけターミネーターはどうでもいい存在だ、ってのが今回の結論ですね。
(ただし、大腸菌に作らせる今回のソーマチン実験みたいな場合は、環状のプラスミドが使われますから、この場合、ターミネーターは断然あった方がいいでしょう。
別に合成されるRNAが長くなっても致命的な問題はないけれど、ダラダラ合成が続くと無駄にエネルギーが必要になって収量も落ちるだろうし、RNAが長くなりすぎると分解もされやすくなったりするでしょうから、あって悪いことも一切ないので、pET-15bにはちゃんとターミネーターが配置されている、って感じですね。)
また、プラスミドを使って、チューブ内でRNAを合成する場合(生物=大腸菌ではなく、mRNAワクチンを作るのと同じように、チューブで合成する場合)は、適当な制限酵素でプラスミドを切って(もちろん、切断部位は、欲しいRNA全長より下流に位置するものです)、直線状にしてから使うことが基本になります(って、先ほどの論文でもまさにそうされていた感じですね)。
ターミネーター配列があっても正直100%は信用なりませんし、制限酵素で切断すれば物理的にそこが絶対終わりになるので安心確実&一箇所切った方がプラスミドDNAの精製などもしやすい、といった理由があるからですが、もしpET-15bを使うなら……まぁマップを見て、T7プロモーターで作られる遺伝子矢印の下流にある、HindIIIとかが使われるでしょうかね。一番安い酵素ですし。
…ってまぁpET-15bはコピーナンバーも小さいので、鋳型となるプラスミドDNAを大量に用意しづらいですから、わざわざこれを使ってチューブ内で転写反応を行うことはまずなされませんが(ちなみに、チューブ内で行う反応をin vitro、生体内で行う反応をin vivoといいます。まぁ結構出てくる重要用語ですけど、入門編としてはどうでもいいでしょう)、理論上は、これを使ってチューブ内で転写を行うことも余裕で可能、って話ですね。
(改めて、普通はin vitro転写でプラスミドを使うならコピーナンバーの大きいプラスミドを使いますし、今の時代ならPCRを使うことも多いです。PCRは、話題のネタともいえますし、またいつか触れたい話ですね。)
…と、ターミネーターについて、あんまりハッキリしないだけの話で長くなっちゃいましたが、次回はようやく、DNA→RNAから一つ進めた、新しいステップの話を見ていくといたしましょう。