引き続き、オペロンとかいう、授業で習わされる割に正直そんなに重要でもない知識を踏まえて、話を進めていきましょう。
前回見ていた通り、大腸菌にはラクトースオペロンと呼ばれる仕組みが存在しており、ラクトース分解酵素遺伝子は、リプレッサーと呼ばれる「転写OFFスイッチ押し係」の力で抑制的に、いわゆる負の制御を受けている、という話でした。
(それのおかげで、リプレッサーを捕まえて構造を変える能力があるラクトースが増えたら「OFFスイッチがOFF」にされて、ラクトース分解酵素の遺伝子がスイッチON→その結果、ラクトースが分解される=「OFFスイッチをOFF」にするやつが減るので、また徐々にOFFスイッチが押されるようになる…という回路のようなフィードバック制御が完成する。)
この中々よくできた仕組みを、今回ソーマチン実験で使おうと紹介しているpETシステムでは採用しているんですね!
そもそも導入した遺伝子のタンパク質を大量に合成したい実験なのに、なぜそんなOFFスイッチなんかが採用されているかといいますと、これは割と単純で、合成したいタンパク質は大腸菌にとっては異物も異物で、ものによっては毒性を発揮することもあるわけです。
ソーマチンみたいにただの甘いタンパク質なら、まぁもしかしたら大腸菌は「うめぇうめぇ(笑)」とエサにできるかもしれませんが(まぁ、タンパク合成用の大腸菌はタンパク質分解酵素をノックアウトして壊しているので、多分消化できませんけどね)、どんなタンパク質を合成させるかは実験次第で、場合によっては大腸菌の生育に不利だったり、それこそ本当に菌そのものを殺してしまいかねない危険なタンパク質を作りたい場合もあるわけです。
(というか、毒性がなくても、大っ量~に自分にとってはよく分からんタンパク質を自分の細胞の中で作らせられたら、どんなものでも不快というか邪魔なのは、何となく想像できるのではないかと思います。)
なので、こういった大腸菌を使ったタンパク質合成実験では、最初の内は遺伝子のスイッチをOFFにして、邪魔なものは作らずに、大腸菌さんにはのびのびと健やかに生育(分裂)して数を増やしていただいて、いざそれなりに増えたら、スイッチをONにして一気に合成スタート!という戦略をとっているわけです。
その、大腸菌培養初期、すなわち菌の数を増やすフェーズで、余計なものが生まれて分裂増殖を邪魔しないようしっかりと導入遺伝子のスイッチをOFFにするために、ラクトースオペロンが(pETシステムの偉い開発者によって)採用されている、という話なんですね。
要は、作りたいものは大腸菌にとって毒(とまではいかなくても、余計なものを作るのは生育に不利)なので、プラスミドをもった菌がちゃんと増えるまではお漏らしを防ぐべく、ストッパーを用意している、という話に他なりません。
具体的には、こないだのこの記事で大腸菌Wikiの情報を見ていた通り、遺伝子スイッチであるT7 RNAポリメラーゼは、(λ(DE3)というファージに感染させて得られたものでしたが、これをより詳しく見ると)lacUV5というスイッチの制御下に置かれたものであって、このlacUV5というのが、まさしくラクトースオペロンに他ならないわけです。
(lacUV5は、オリジナルのラクトースオペロンと比べて、2塩基ほど変異が入っている(=RNA合成開始点から10塩基ほど上流の、TATGTTが、TATAATに変わっている)もので、オリジナルのプロモーターよりRNA合成量がアップしているというナイスな特徴をもっています。)
つまり、生育初期・分裂して菌がいっぱい増えている段階では、リプレッサーがlacUV5のオペレーター部位に結合して、T7 RNAプロモーターの合成を邪魔しているという仕組みなわけですね。
そういえば今更ですが、そのリプレッサーがどういうものなのか、また「こんなの見ても一切何も分からない」で有名な、結晶構造の図だけ申し訳程度に引用しておくとしましょう。

こちらがラクトースオペロンで使われるlacリプレッサーで、遺伝子名はLacIとなっていますが(「なんでI(アイ)なんだよ!リプレッサーならRじゃねぇのか!」とも思えますが、まぁリプレッサーはあくまで一般名なので、物質名とは一致しなくても仕方ないのかもしれませんね。このIは、恐らくinhibit(抑制する)という生命科学でめちゃくちゃよく使われる単語由来ではないかと思いますが、ソース付きでそう明示している情報は見当たりませんでした)、まぁ一応、情報量ゼロとうわけではなく、それなりに特徴は描かれています。
トップの金色がDNAのオペレーター領域で、LacI上部のDNA結合部位(binding domain)がそことガッチリ結合しているわけですけど、これも(こないだ見ていた、6個集まって機能するラージT抗原同様)LacIというタンパク質1分子が4つ集まって機能する四量体として存在している(画像には2つ=二量体が表示されているだけですが、二量体が2つ揃って機能するようです。2つの二量体は、それぞれ異なるオペレーター領域を認識することも知られているみたいですね)ようですけれども、まぁそんなこたぁどうでもいいでしょう。
画像下半分の、調節部位(Regulatory domain)にラクトースが結合すると、DNA結合部位の構造がガラッと変わって、オペレーター部位とは結合できなくなってしまう、というのが前回見ていた話のおさらいですね。
構造はともかく、このLacIは、一体どうやって作られているのか?
これは、もともとラクトースオペロンというのは大腸菌自身のもつメカニズムですし、大腸菌自身が作ることもできるわけですが、その量はとても少なく、しっかり制御するには足らないようなので、追加で作らせています。
そう、先ほども見ていたこないだの記事(&大腸菌Wiki)に載っていたとおり、大腸菌に感染させたDE3には、T7RNAP遺伝子のみならず、lacI遺伝子も乗せてあるんですね。
(再掲)BL21(DE3)の遺伝子型:λ(DE3 [lacI lacUV5-T7p07 ind1 sam7 nin5])
しかも、Wikiの説明によると、このlacIはlacIqと呼ばれる、これもオリジナルから1塩基が変異したパワーアップ版で、LacIがかなりがっつり合成されるようになった強化タイプという形ですね。
なお、リプレッサーLacI自身の遺伝子スイッチは、大腸菌のもつ普通のポリメラーゼが使われているので、これは特に何の制御もなく、常にダラダラと作られる感じですね。
(lacIqの場合、プロモーター部位のちょっとした変異により、ポリメラーゼが結合しやすくなって、より沢山ポコポコ作られている、ということです。)
…という所で一応役者は揃いましたが、うーん、これは正直、とにかくスイッチを制御するためのスイッチがあったり、大腸菌だとかファージだとかプラスミドだとか色々なものが別々の所にあったりで、ハッキリいって分かりづら過ぎる!
しかも、まとめる前に実は更に事態はややこしくなりまして、なんと、リプレッサーによるお漏らし制御を完全なものにするためか、実はプラスミドpET-15bにもlacI遺伝子が存在しています。
プラスミドマップを改めて掲載しましょう。
(こちらはpETシステム開発元のNovagenのマニュアル由来ですが、Novagen自体が既に消滅(メルク・ミリポアに吸収)して公式には手に入りにくくなっていることからも、有志が至る所で公開している画像ですね。既に何度か引用していますが、今回は、環状のマップと配列詳細図をまとめてスクショしたかったので、アクセスしやすかったプラスミドマニア団体・Addgeneから改めて引っ張ってきました。)
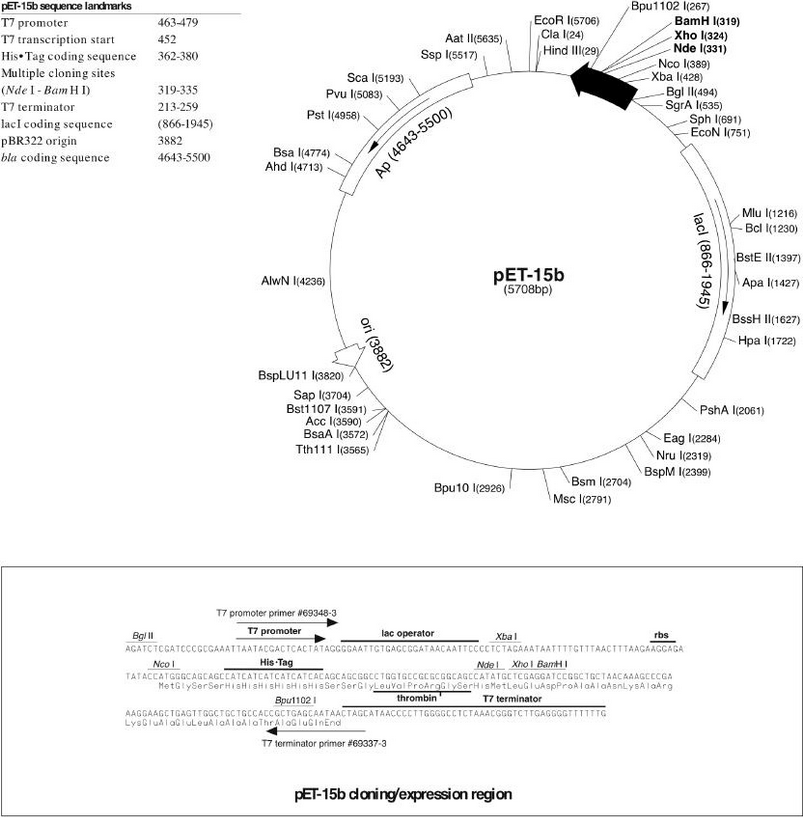
環状マップ、右側ちょうど時計でいうと2時から4時ぐらいの位置に、これまで触れたことはほぼありませんでしたが、実はlacIというのが堂々と存在していたのです。
配列を確認したらこれはパワーアップ版のlacIqではなかったようですが、いずれにせよ恐らくDE3で導入されたlacIqにさらに追加でより沢山のリプレッサーを作る目的、あるいはむしろ、自分の使う菌株によっては大腸菌自身にはlacI遺伝子が存在しないこともありますから、そういうときのために、プラスミドにもちゃんともたせているって意味もあるのでしょう。
そして更にポイントとして、画像下の詳細配列、これは環状マップ黒塗りの矢印の辺の拡大図ですが、T7 RNAポリメラーゼが結合するT7プロモーターの下流すぐに、よく見てみると何と!ここにもlacオペレーターが存在するじゃあありませんか!!
何気に、pETシステムでは、念には念を入れて、T7スイッチにもラクトースオペロンのオペレーター(強制OFFスイッチ)を導入しているんですね。
そこまで厳密にコントロールせにゃあアカンの?とも思えますが、本当に毒性の強いタンパク質を作りたいときは、実際結構きっちり厳密に、お漏らしゼロで菌体を増やしていかないと大腸菌がすぐ死んで実験が失敗する、という話も聞いたことがあります(僕が使ったことのあるタンパク質は、幸いそこまで毒性がなかったので、そんなにこだわるポイントにも思えませんでしたが)。
ということで、何重にもスイッチのスイッチのスイッチみたいな感じで入り組んでいるpETシステムですが、改めてじっくりおさらいしておくとしましょう。
これ、下流から見ていった方が分かりやすいのかなぁ…。
流れとしては逆ですが、まずは下流にあたる部分から箇条書きでポイントを抑えていくとしましょうか。
- 目的の遺伝子の転写(DNA→RNAの変換)は、T7 RNAポリメラーゼがT7プロモーターに結合することで行われる(=目的遺伝子のスイッチON)
- ただし、T7 RNAポリメラーゼは、LacIがオペレーター領域に結合して邪魔していると働けない(=目的遺伝子の強制スイッチOFF)
- 一方そのT7 RNAポリメラーゼ自体は、lacUV5プロモーター(λDE3ファージを感染させて、大腸菌ゲノムに導入した組換え遺伝子)にRNAポリメラーゼ(これは大腸菌自身が元々もつ、名もなき普通のRNAポリメラーゼ)が結合することで作られる(=T7 RNAポリメラーゼ遺伝子のスイッチON)
- ただしこれも、このlacUV5プロモーター下流にはlacオペレーターがあるので、LacIが存在するとブロックされる(=T7 RNAポリメラーゼ遺伝子の強制スイッチOFF)
- そのLacIは、これも大腸菌自身のRNAポリメラーゼを使って、組換え大腸菌のもつlacIq遺伝子(λDE3ファージの感染で獲得)およびpET-15bに存在するlacI遺伝子から、じゃんじゃか作られる(=lacI遺伝子のスイッチ、これはいわば常時ON)
- (なおちなみに、「大腸菌自身のRNAポリメラーゼ」も、大腸菌が生きるために必要なものなので、これは大腸菌自身の遺伝子DNAから沢山作られている(常に必要なので、特にオペロン制御とかはなく、常時作られて細胞内に豊富にある感じ))
…という感じで、改めて上流から見直してみると……
・リプレッサーであるLacIが沢山作られている
→T7 RNAポリメラーゼ遺伝子がOFFになるのでT7 RNAポリメラーゼが作られない
(=目的遺伝子のスイッチはONにならない)
→仮にちょっとお漏らししてT7 RNAポリメラーゼができちゃったとしても、目的遺伝子のスイッチ自体もLacIによりOFFにされているので、T7 RNAポリメラーゼは機能できない
⇒目的遺伝子は、二重ブロックで、かなりしっかりとOFFに制御されている!
…といった具合に、スイッチを意図的にONにするまでは、結構厳密に目的遺伝子のタンパク質が作られちゃわないように設計されている、よくできたメカニズムになってるってことですね。
(正直、逆向きに見る意味もあんまなかった気もしますが(笑)。
…多分、そんな見る順番うんぬんではなく、各種遺伝子が、「別の遺伝子をONにする物質」として働くことも、「自分自身がONにされる対象」として出てきていることもある…というそのややこしさが、整理&理解するうえで一番の難関ポイントかな、という気がします。)
では、これをスイッチONにするにはどうするのか?
それこそが次のステップで、ある程度まで大腸菌の数を増やしたら、満を持して、リプレッサーを引っぺがしてタンパク質を大量に合成し始めてもらう感じなわけですね。
無駄に引っ張ってる感じになっちゃってますが、それについての詳細は次回見ていくとしましょう。