DNAシーケンシング反応の話で、説明不足で片手落ちになっていた部分の補足をここ何回かやらせてもらっていました。
いただいたご質問に答える形で、早速続きに参りましょう。
Q8. 「ゲルを流してなんちゃらする」という作業が、どうにもイメージが湧かない。一体、何がどうなっているのか?!
A8. ゲルを使った実験、あまりにも常日頃やりすぎていて、自分にとって当たり前なことになりすぎていた結果、完全に説明が欠けてしまっていましたね…!
こんなもん聞かなきゃイメージできるわけもありませんから、せっかくなのでちょっと詳しく触れてみるとしましょう。
まず生命科学研究で日常的に使われるゲルには主に2種類あって、名前だけはどちらも出したことがあるんですけど、アガロースゲルと、「タンパク質を解析する実験としてSDS-PAGEというのがあります」と語っていたこのPAGEの「PAG」の部分、ポリアクリルアミドゲルというものの2つになります(PAGEは、polyacrylamide gel electrophoresisで、そのまんま、「ポリアクリルアミドゲル電気泳動」の頭字語です)。
使い分けとしては、DNAがアガロースゲル、タンパク質がPAGE……であることが多いので最初はそう思うんですが、別にそういう決まりはなく、単純に、より大きい分子を流すのにはアガロースが適していて、より小さい分子を流す&膜に移す(こないだ見ていたブロッティングですね)目的ではPAGEがより適しているというだけで、PAGEでDNAを流すことも普通にあります。
(まぁ、タンパク質をアガロースゲルで流すことは、絶対にないわけではないでしょうが、僕はやったことがないですけどね。
DNAは設計図であり、遺伝子のスイッチ領域なども含んでいるので巨大になりがち(そもそもDNA3文字でアミノ酸1つですしね)で、染色体はもちろん、プラスミドを見ても色々なエレメントが1つのリングに乗っていたことなんかからも分かる通り、DNAとタンパク質を比べたら、DNAの方がより大きいことがほとんどなんですね。
タンパク質の場合、そんなに巨大な分子を使うことはまずないので、大きい分子用のアガロースが必要になることはまずない、というわけです。)
まぁそういう細かすぎてどうでもいい点はともかく、もっと大切な、原理というか仕組みというか、そもそもこいつらはどんなもので、何をして何を見とんねん、という話をしていきましょう。
結局、アガロースというのは寒天みたいなものなのです。
一方、ポリアクリルアミドは、これはゼリーですね。厚さ1 mmとかの、薄~く伸ばして板状になった、プルプルした食感の透明のゼリーです(まぁ食べはしませんけど(笑))。
そもそもゲルというのは、あんまり日常生活でなじみのある語じゃないかもしれないですけど、ドロッとしたスライムのような、弾性のある固形物質全般を指す言葉ですが、ジェルといえばよりイメージが湧く感じでしょうか。
ゲルとジェルは、日常会話ではもしかしたら微妙に違うニュアンスで使われる言葉かもしれないですけど(ハンドクリームジェルとか、ヘアジェルとか、スライムっぽいけど、固体より液体に近いイメージ?)、実は両者は全く同じ、gelという物質になります。
英語読みでジェル、ドイツ語読みでゲルですね。
ということでゲルというのはジェルをイメージしてもらえばいいわけなんですけど、まぁ上述の通り、一般的な「ジェル」と聞いて抱くイメージより、もっとしっかりした固体、まさに寒天とか、フルーツゼリーみたいなものの方が、実験で使うゲルには近い感じですね。
このゲルという物質は、弾力がありプルプルした感触の物質なんですけど、顕微鏡とかで超拡大して見てみると、つまりミクロのレベルでは、網目構造をもっていることがその最大の特徴になります。
要は、寒天みたいな四角いゲルは、小さい穴(孔と書いた方が正確かもしれませんが)の開いたネットのようなものが三次元的に張り巡らされている物質だ、といえるわけですね(弾力があるのも、そのことに起因するわけです)。
そして、この網目構造をもっているが故に、ちょうどネットの隙間を通れるぐらいのサイズである分子を、その大きさごとに分けることができるわけです。
高分子学会の解説記事にあった、この画像がちょうど分かりやすい感じでしょうか。
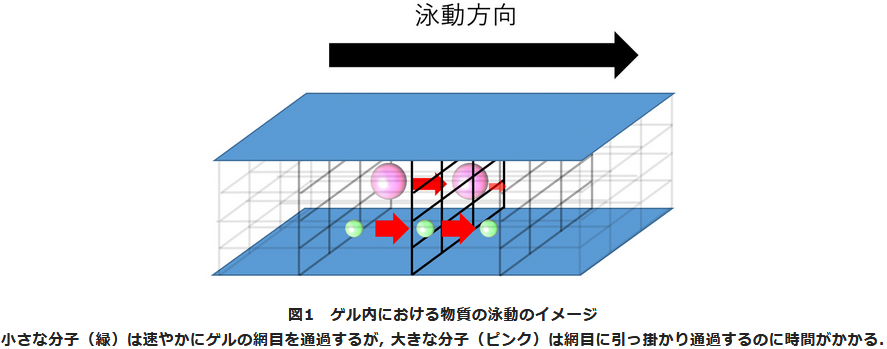
小さい分子は網目に引っかかりにくく、スルスルと移動できる……一方、大きい分子はネットにガンガン引っかかるので、モタモタとかなり移動が遅くなる……という、ズバリいってそれだけの話になるんですね(大きければ大きいほど、引ってかりが増えて移動度が小さくなる)。
これを、あんまり専門用語っぽくない専門用語で分子ふるい効果といいますが、まぁそんな用語はともかく、この効果は絶大で、出発地点から分子を同じ方向に同じスピードで移動させると、移動度の違いで、確実に、正確に、小さい順に分子を並べることが可能になるのです。
「えぇ~、そんな上手いことみんな網目に引っかかるのぉ?たまたま網目が粗い所とかがあって、そこだけ大きいやつも速く動けた、みたいなことはないわけぇ?」みたいなそういう「そんなランダムな引っかかりに依存する話で、バラつきはないのか?」という気も若干するかもしれないんですけど、まぁ寒天とかは不純物も多いのでそういう心配もあるかもしれないものの、そのために、電気泳動のゲルには、たっかいたっかい精製されたアガロースを使っているわけですしね。
純品からなるゲルは非常に均一で、実際僕自身もう何万回もゲルを流してますけど、本っ当~に見事に、完全に理論通り、キレーイに分子が大きさ順に一列に並びます。
(そういえばよく「ゲルを流す」といっちゃいますが、正確には「ゲルでDNAを流す」という使い方をすべき用語ですね。まぁ雰囲気で通じると思うので適当に書いちゃってますけど、当然、ゲル自体は流れません(笑)。固定したゲルに、DNAやタンパク質といった分子を流す、ということですね。)
ちなみに流し方は、もう何度も書いてますけど、電気を使って流します。
DNAは分子の構造的に、マイナスの電荷を帯びている(リン酸基が負に帯電しているため)ので、電気を流すと、陽極に向かって流れていくわけですね。
タンパク質は、構成するアミノ酸によって分子自体がもつ電荷はマチマチなので、SDSをからめることで、構造をほどいてやるとともに、強制的に負に帯電させるという形になっています(ややこしい話なので、特に気にしなくてよいでしょう)。
要は、ゲルの端にサンプルを乗せる穴ぼこを開け、そこにサンプルを注入して、電気を流せば、分子はみんな陽極に向かってゲルの中を走り始め、網目の大きさに応じてでかいやつは引っかかりまくって遅く、小さいやつはスルスルと移動できて速く移動していく…というのが電気泳動でやってることの全てなわけですね。
アガロースが大きい分子用、ポリアクリルアミドが小さい分子用というのはまさに網目のサイズによるもので、アガロースは割とスカスカ、一方ポリアクリルアミドはかなり密な目を形成しているのです。
なので、タンパク質みたいな小さいものをアガロースゲルで流すと、全員スイスイと移動してしまって上手く分けることができないし(全部がほぼ引っかかりなしで最速で移動できてしまうので、ただ先端に集まるだけ)、一方、巨大なDNAをPAGEで流すと、全員網目に引っかかりすぎて全く移動できず、出発点に留まり続けるだけでこれまた全然分離ができない、ということになるんですね。
よって、使うゲルは、見たい分子の大きさに応じて使い分ける必要があるわけです。
(同じアガロースでも、より巨大なDNAをしっかり分けたいときはアガロース0.6%濃度といったかなりヤワいゲル、逆に、より小さめのDNAをキッチリ分離したいなら、2%ゲルとかいったかなりカッチカチのゲルを使う…のように、濃度を変えて使い分けることもしばしばです。)
仕組みが分かった所で、現物を見てみましょう。
アガロースゲルは、恐らく日本中ほとんどの研究室が、Mupid(ミューピッド)と呼ばれるゲルメーカーを使って作っていると思います。
まぁゲルメーカーといっても全く大したものではなく、トレイと、穴あけ用のクシ(コーム)があるだけなんですけどね。
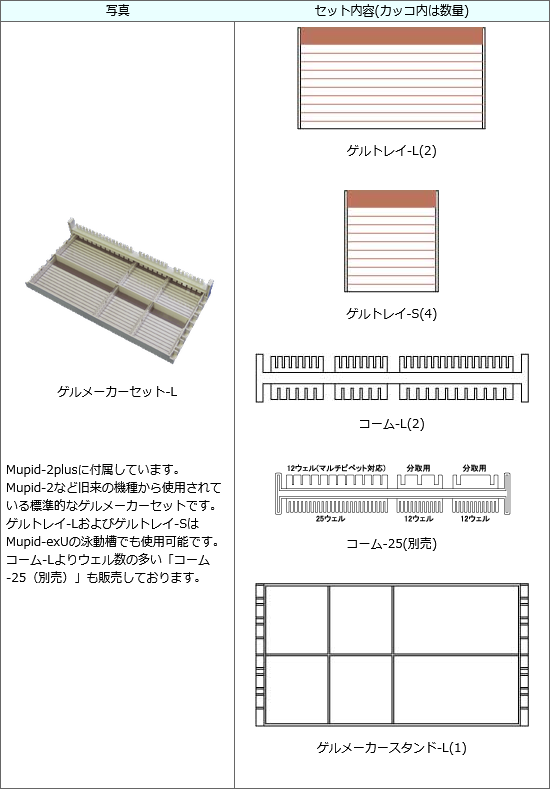
必要な量のアガロースを水に溶かし(水は電気が流れにくいので、緩衝液と呼ばれる塩水の一種ですが)、レンジでチンして均一に溶かした後、固まる前にトレーに注いで、後は冷めて固まるのを待つだけになります。
先ほどの高分子学会の記事にもちょうどミューピッドを使った実験の実例が掲載されていましたが、コームで開けた穴(ウェルと呼んでいます)にDNAサンプルを沈めて(ウェルの中に沈むように&肉眼で見て泳動されていることが分かるように、サンプルには比重の大きい色素を混ぜます)、電気を流すだけですね。

ミューピッドは日本が誇る素晴らしいゲルメーカーのようで、海外から来た研究者の方は絶賛するらしいですが(実際、学生の頃の先輩に海外から来た大学院生がいましたが、卒業して帰るとき、「ミューピッド持って帰りてぇー!」っていってた気がします)、ぶっちゃけ、「は?こんなんの何が特別なん?誰でも思いつく、ただのプラスチック容器じゃん」と思える気もするのが正直な所かもしれません…。
…が、実際海外の研究室で使われるゲルメーカーは、確かに、何か泳動タンク(先ほどの高分子学会の実験例の図)のゲルを乗せる台の両端にストッパーを付けて、そこで固めるみたいな柔軟性の全くないクソ仕様のものばかりで(ストッパーがゆるいと、固まる前にゲルが漏れるみたいな)、確かにミューピッドの方が使いやすいな、というのは実際かもしれません(何だかんだ、僕もこっちに来てミューピッドを買いましたしね。届いた説明書が日本語だけで、「いや海外の人読めんやん!」と笑えた記憶があります)。
一方PAGEの方は、ゲルが薄いということもあり、作製に若干手間がかかります。
こちらは、(多少製品によって違いはあるものの)ガラス板2枚の間にスペーサーを挟み、クリップで固定して正立させ、さらにタンパク質用のSDS-PAGEの場合、伝統的に二層のゲルを作るという、「レンチン→注ぐだけ」のアガロースより、やや面倒くさい手順になっている感じです。
相変わらず言葉だけの説明は分かりにくいですね。
電気泳動関連製品大手のATTOに、写真や動画まであったので、参考に引用させていただきましょう。

サイトにはゲル作製の各ステップが写真つきで掲載されていますが、それをまとめた動画が一番分かりやすいかもしれませんね(まさかの無音・紙芝居動画でしたが(笑))。
アクリルアミドのゲル化は、温度変化ではなく、化学的な構造変化で行います。
アクリルアミドの単量体(モノマー:水に溶かせば、完全な液体)に、特別な試薬(この記事で触れていた、不快臭物質TEMEDなど)を加えて、ズラーっとつなげてポリマーにする(高分子化して、ゼリーのようになる)形ですね。
まぁ作り方はどうでもいいんですけど、こんな感じの、以前いっていた通り大体スマホサイズぐらい(もうちょい大きいかな?)の透明で薄っぺらいものが、PAGEで使うゲルという感じです。
これを、泳動槽にセットして、アガロースの場合と同様、電気をかけてサンプル分子を流す形になります。
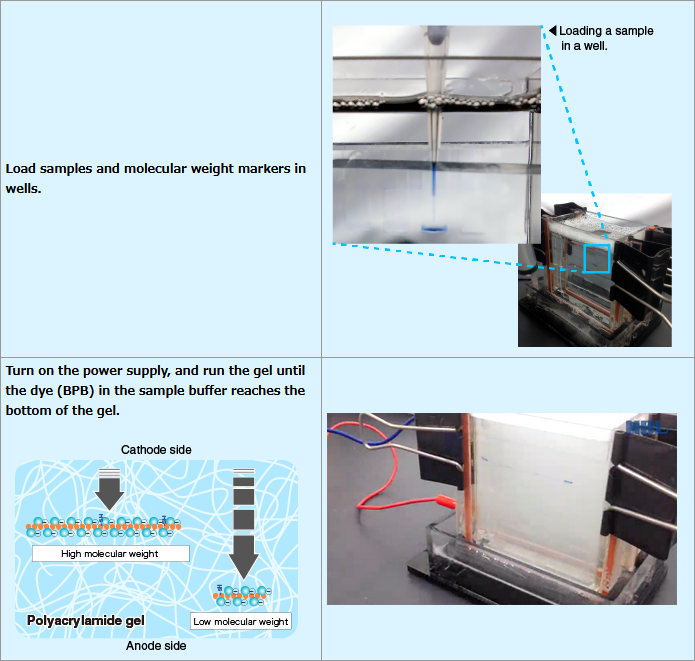
ゲル製作後、実際にサンプルを流している様子のいい写真があまりなかったのですが、まぁこんな感じ(↓)で、電気を通して上から下に流す感じですね。
(結構高圧をかけるので、泡が発生するし、ゲルも熱を帯びます。)
そういえばアガロースゲルは水平に流す一方、PAGEは垂直に流すのが世界中どこでもデフォルトになっていますが、何でなんでしょうね?
恐らく、PAGEはゲルが薄いから、寝かせて使うのが難しい(=サンプル乗せ用のウェルを、ゲルの厚みの部分に作るのが難しい。上記動画にもあった通り、PAGEでは、ゲルを泳動の進行方向に掘ってウェルを作る形ですね)、ってのがある気がしますが、まぁ水平アクリルアミドもなくはないと思うので、必ずしも絶対そうではないのかもしれません。
作るのも、使うのも、全体的にアガロースの方が楽チンですが、PAGEの方が網目も細かいので分解能・解像度が高い&シャープなバンドが得られるというメリットはあるといえそうです(もちろん、巨大分子の泳動には向かない(全くゲルに入っていかないので)のは前述の通りですが)。
…と、そんな感じで、ゲル電気泳動というのは行われるという話でした。
これで、サンガーさんの開発した「ゲルに流して1塩基ずつの違いを見る方法」が何をやっていたのかも、ご理解いただけるのではないかと思います。
(古典的なサンガー法の例、再掲)

ちなみに、アガロースゲルで1塩基単位の違いを見るのは不可能なので(アガロースは、大きな分子用と書いた通り、例えば500塩基のインサートと5000塩基のベクターの差を見るというのには適していますが、「16塩基と17塩基の違いを見る」とかには向いていないというか絶対に不可能です)、これはPAGEで見ることになる感じですね。
アクリルアミド濃度の高いPAGEなら、プライマーが1塩基伸びた16塩基にはじまり、そこから1塩基ずつの違いも十分検出可能です。
上の図には描いていませんでしたが、ウェルが上方にあり、4つのウェルにそれぞれの反応溶液を乗せて、上から下に電気を流す(下が陽極で、DNAは下に向かって移動する)ということですね。
DNA分子たちは非常に素直で、確実に、例外なく、キッチリと小さいものから順番に速く移動していき、最終的にこういうハシゴ状の泳動パターンが得られる、ということでした。
なお、蛍光ddNTPを使った反応の方は、1つのチューブに入れて一気に流せるわけですが、PAGEよりも更に分離能も解像度にも優れた、キャピラリーを用いる、という風に書いていました。
キャピラリーは、日常的に各研究室で気安く使うタイプのものではなく、シーケンシング装置(シーケンサー)とかそういう大層な機械でのみ使われるやつで、僕も学生時代にシーケンサーを使っていたときに触ったことはありますけど、それ以外で使ったことはありません。
…が、原理は全く同じ、「小さいものが速く移動するので、キャピラリーの先端にある蛍光検出器で、流れてきたDNA断片の色を順番に記録していく」というだけの形ですね。
無駄に長い図ですが、こちらも再掲しておきましょう。
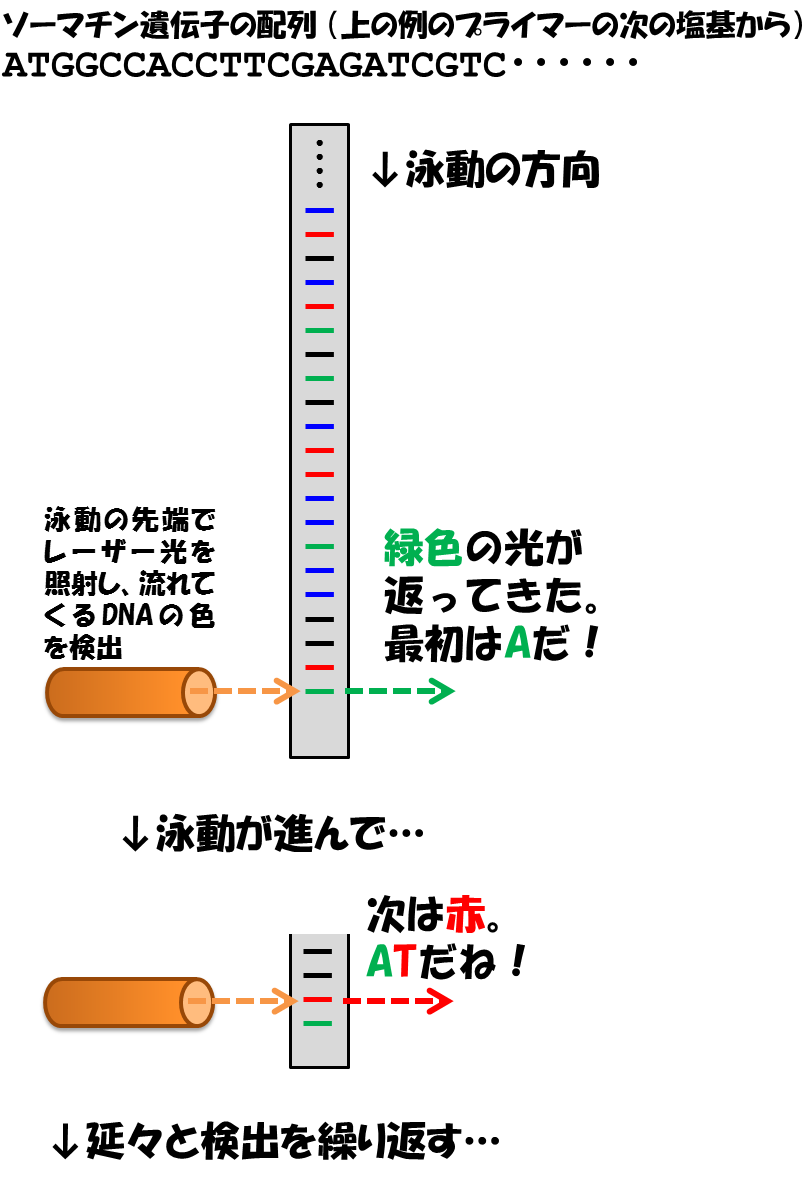
グレーの四角が、この場合はゲルではなくキャピラリー(キャピラリーの中にはゲルが詰まっているのでまぁゲルともいえますけど、多分アクリルアミドよりもっと分解能に優れた特殊なポリマーが充填されてるんだと思います)ですね。
電気の力で大きさ順に流れてくる蛍光付きDNA断片を、順番に検出していくという、よくできたマシーンです。
といったところで、シーケンシング反応とその検出については、概ね説明が済んだかと思います。
次回は、次世代シーケンス……は正直複雑すぎて、これまで以上にややこしくてイミフになり、書く意味がないのでどうかな、と思ったんですけど、仕組みに深入りしない範囲で概略ぐらい見ていこうかな、などと思っとります。